3D图像分类从CT扫描
作者: Hasib Zunair
创建日期: 2020/09/23
最后修改: 2024/01/11
描述: 训练3D卷积神经网络以预测肺炎的存在。
介绍
本示例将展示构建3D卷积神经网络(CNN)以预测计算机断层扫描(CT)中病毒性肺炎存在所需的步骤。2D卷积神经网络通常用于处理RGB图像(3个通道)。3D卷积神经网络则是3D的等价物:它接受3D体积或2D帧序列(例如CT扫描中的切片)作为输入,3D卷积神经网络是学习体积数据表示的强大模型。
参考文献
设置
import os
import zipfile
import numpy as np
import tensorflow as tf # 用于数据预处理
import keras
from keras import layers
下载MosMedData:带有COVID-19相关发现的胸部CT扫描
在本示例中,我们使用 MosMedData:带有COVID-19相关发现的胸部CT扫描的一个子集。 该数据集由肺部CT扫描组成,其中一些具有COVID-19相关发现,另一些则没有这些发现。
我们将使用CT扫描的相关放射学发现作为标签来构建分类器,以预测病毒性肺炎的存在。 因此,这个任务是一个二分类问题。
# 下载正常CT扫描的URL。
url = "https://github.com/hasibzunair/3D-image-classification-tutorial/releases/download/v0.2/CT-0.zip"
filename = os.path.join(os.getcwd(), "CT-0.zip")
keras.utils.get_file(filename, url)
# 下载异常CT扫描的URL。
url = "https://github.com/hasibzunair/3D-image-classification-tutorial/releases/download/v0.2/CT-23.zip"
filename = os.path.join(os.getcwd(), "CT-23.zip")
keras.utils.get_file(filename, url)
# 创建一个目录以存储数据。
os.makedirs("MosMedData")
# 解压数据到新创建的目录中。
with zipfile.ZipFile("CT-0.zip", "r") as z_fp:
z_fp.extractall("./MosMedData/")
with zipfile.ZipFile("CT-23.zip", "r") as z_fp:
z_fp.extractall("./MosMedData/")
正在从 https://github.com/hasibzunair/3D-image-classification-tutorial/releases/download/v0.2/CT-0.zip 下载数据
1045162547/1045162547 ━━━━━━━━━━━━━━━━━━━━ 4s 0us/step
加载数据和预处理
文件以Nifti格式提供,扩展名为.nii。为了读取扫描,我们使用nibabel包。
您可以通过pip install nibabel安装该包。CT扫描以Hounsfield单位(HU)存储原始体素强度。
在该数据集中范围从-1024到2000以上。
超过400的是具有不同射线强度的骨头,因此这被用作上界。通常使用-1000和400之间的阈值来规范化CT扫描。
为了处理数据,我们执行以下操作:
- 我们首先将体积旋转90度,以固定方向
- 我们将HU值缩放到0到1之间。
- 我们调整宽度、高度和深度。
在这里,我们定义几个辅助函数来处理数据。这些函数 将在构建训练和验证数据集时使用。
import nibabel as nib
from scipy import ndimage
def read_nifti_file(filepath):
"""读取并加载体积"""
# 读取文件
scan = nib.load(filepath)
# 获取原始数据
scan = scan.get_fdata()
return scan
def normalize(volume):
"""标准化体积"""
min = -1000
max = 400
volume[volume < min] = min
volume[volume > max] = max
volume = (volume - min) / (max - min)
volume = volume.astype("float32")
return volume
def resize_volume(img):
"""沿z轴调整大小"""
# 设置所需深度
desired_depth = 64
desired_width = 128
desired_height = 128
# 获取当前深度
current_depth = img.shape[-1]
current_width = img.shape[0]
current_height = img.shape[1]
# 计算深度因子
depth = current_depth / desired_depth
width = current_width / desired_width
height = current_height / desired_height
depth_factor = 1 / depth
width_factor = 1 / width
height_factor = 1 / height
# 旋转
img = ndimage.rotate(img, 90, reshape=False)
# 沿z轴调整大小
img = ndimage.zoom(img, (width_factor, height_factor, depth_factor), order=1)
return img
def process_scan(path):
"""读取并调整体积大小"""
# 读取扫描
volume = read_nifti_file(path)
# 标准化
volume = normalize(volume)
# 调整宽度,高度和深度
volume = resize_volume(volume)
return volume
让我们从类别目录中读取CT扫描的路径。
# 文件夹 "CT-0" 包含正常肺组织的CT扫描,
# 没有病毒性肺炎的CT迹象。
normal_scan_paths = [
os.path.join(os.getcwd(), "MosMedData/CT-0", x)
for x in os.listdir("MosMedData/CT-0")
]
# 文件夹 "CT-23" 包含具有多种毛玻璃样模糊的CT扫描,
# 肺实质的受累。
abnormal_scan_paths = [
os.path.join(os.getcwd(), "MosMedData/CT-23", x)
for x in os.listdir("MosMedData/CT-23")
]
print("具有正常肺组织的CT扫描数量: " + str(len(normal_scan_paths)))
print("具有异常肺组织的CT扫描数量: " + str(len(abnormal_scan_paths)))
具有正常肺组织的CT扫描数量: 100
具有异常肺组织的CT扫描数量: 100
构建训练和验证数据集
从类别目录中读取扫描并分配标签。对扫描进行下采样,使其形状为128x128x64。将原始HU值重新缩放到0到1的范围内。最后,将数据集拆分为训练和验证子集。
# 读取和处理扫描。
# 每个扫描在高度、宽度和深度上进行调整,并进行重新缩放。
abnormal_scans = np.array([process_scan(path) for path in abnormal_scan_paths])
normal_scans = np.array([process_scan(path) for path in normal_scan_paths])
# 对于具有病毒性肺炎存在的CT扫描
# 分配1,对于正常的分配0。
abnormal_labels = np.array([1 for _ in range(len(abnormal_scans))])
normal_labels = np.array([0 for _ in range(len(normal_scans))])
# 按70-30的比例拆分数据,用于训练和验证。
x_train = np.concatenate((abnormal_scans[:70], normal_scans[:70]), axis=0)
y_train = np.concatenate((abnormal_labels[:70], normal_labels[:70]), axis=0)
x_val = np.concatenate((abnormal_scans[70:], normal_scans[70:]), axis=0)
y_val = np.concatenate((abnormal_labels[70:], normal_labels[70:]), axis=0)
print(
"训练和验证样本的数量分别为%d和%d."
% (x_train.shape[0], x_val.shape[0])
)
训练和验证样本的数量分别为140和60.
数据增强
CT扫描在训练期间通过随机旋转进行增强。由于数据存储在形状为(样本, 高度, 宽度, 深度)的三维张量中,我们在轴4添加大小为1的维度,以便能够对数据执行3D卷积。因此,新的形状为(样本, 高度, 宽度, 深度, 1)。有不同种类的预处理和增强技术,这个示例展示了一些简单的方法以便入门。
import random
from scipy import ndimage
def rotate(volume):
"""将体积旋转几个度数"""
def scipy_rotate(volume):
# 定义一些旋转角度
angles = [-20, -10, -5, 5, 10, 20]
# 随机选择角度
angle = random.choice(angles)
# 旋转体积
volume = ndimage.rotate(volume, angle, reshape=False)
volume[volume < 0] = 0
volume[volume > 1] = 1
return volume
augmented_volume = tf.numpy_function(scipy_rotate, [volume], tf.float32)
return augmented_volume
def train_preprocessing(volume, label):
"""通过旋转和添加通道处理训练数据。"""
# 旋转体积
volume = rotate(volume)
volume = tf.expand_dims(volume, axis=3)
return volume, label
def validation_preprocessing(volume, label):
"""仅通过添加通道处理验证数据。"""
volume = tf.expand_dims(volume, axis=3)
return volume, label
在定义训练和验证数据加载器时,训练数据通过增强函数,随机旋转体积。注意训练和验证数据已经缩放到0到1之间的值。
# 定义数据加载器。
train_loader = tf.data.Dataset.from_tensor_slices((x_train, y_train))
validation_loader = tf.data.Dataset.from_tensor_slices((x_val, y_val))
batch_size = 2
# 在训练期间动态增强。
train_dataset = (
train_loader.shuffle(len(x_train))
.map(train_preprocessing)
.batch(batch_size)
.prefetch(2)
)
# 仅进行缩放处理。
validation_dataset = (
validation_loader.shuffle(len(x_val))
.map(validation_preprocessing)
.batch(batch_size)
.prefetch(2)
)
可视化一个增强后的CT扫描。
import matplotlib.pyplot as plt
data = train_dataset.take(1)
images, labels = list(data)[0]
images = images.numpy()
image = images[0]
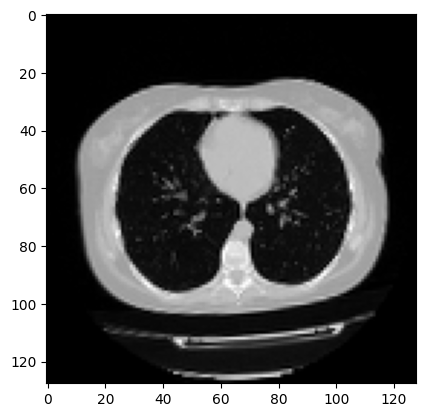
print("CT扫描的维度是:", image.shape)
plt.imshow(np.squeeze(image[:, :, 30]), cmap="gray")
CT扫描的维度是: (128, 128, 64, 1)
<matplotlib.image.AxesImage at 0x7fc5b9900d50>
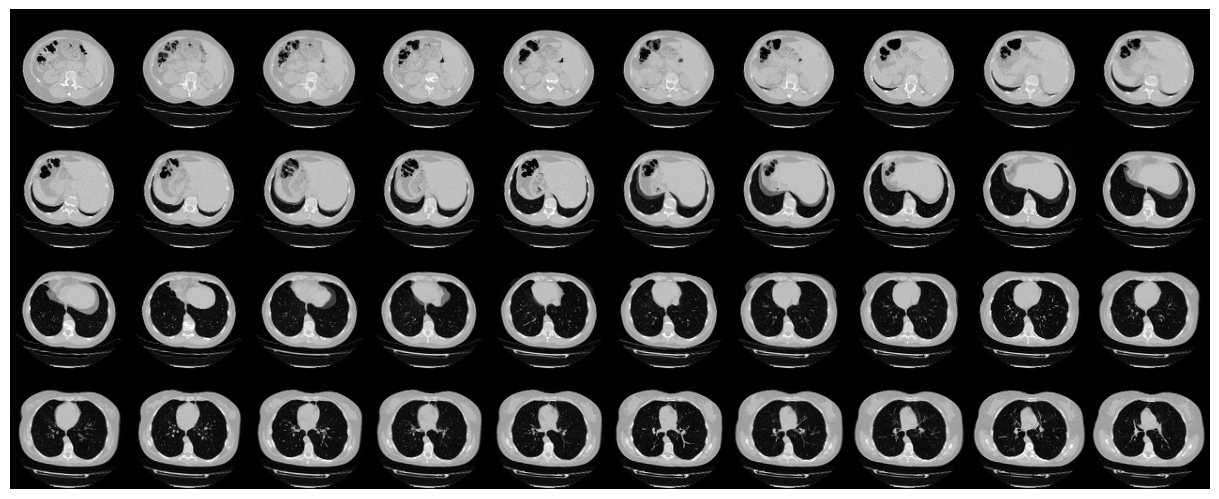
 Since a CT scan has many slices, let's visualize a montage of the slices.
Since a CT scan has many slices, let's visualize a montage of the slices.
def plot_slices(num_rows, num_columns, width, height, data):
"""绘制20个CT切片的蒙太奇"""
data = np.rot90(np.array(data))
data = np.transpose(data)
data = np.reshape(data, (num_rows, num_columns, width, height))
rows_data, columns_data = data.shape[0], data.shape[1]
heights = [slc[0].shape[0] for slc in data]
widths = [slc.shape[1] for slc in data[0]]
fig_width = 12.0
fig_height = fig_width * sum(heights) / sum(widths)
f, axarr = plt.subplots(
rows_data,
columns_data,
figsize=(fig_width, fig_height),
gridspec_kw={"height_ratios": heights},
)
for i in range(rows_data):
for j in range(columns_data):
axarr[i, j].imshow(data[i][j], cmap="gray")
axarr[i, j].axis("off")
plt.subplots_adjust(wspace=0, hspace=0, left=0, right=1, bottom=0, top=1)
plt.show()
# 可视化切片的蒙太奇。
# 4行10列,用于100个CT扫描切片。
plot_slices(4, 10, 128, 128, image[:, :, :40])
定义一个3D卷积神经网络
为了使模型更容易理解,我们将其结构化为块。 这个示例中使用的3D CNN的架构 是基于这篇论文。
def get_model(width=128, height=128, depth=64):
"""构建一个3D卷积神经网络模型."""
inputs = keras.Input((width, height, depth, 1))
x = layers.Conv3D(filters=64, kernel_size=3, activation="relu")(inputs)
x = layers.MaxPool3D(pool_size=2)(x)
x = layers.BatchNormalization()(x)
x = layers.Conv3D(filters=64, kernel_size=3, activation="relu")(x)
x = layers.MaxPool3D(pool_size=2)(x)
x = layers.BatchNormalization()(x)
x = layers.Conv3D(filters=128, kernel_size=3, activation="relu")(x)
x = layers.MaxPool3D(pool_size=2)(x)
x = layers.BatchNormalization()(x)
x = layers.Conv3D(filters=256, kernel_size=3, activation="relu")(x)
x = layers.MaxPool3D(pool_size=2)(x)
x = layers.BatchNormalization()(x)
x = layers.GlobalAveragePooling3D()(x)
x = layers.Dense(units=512, activation="relu")(x)
x = layers.Dropout(0.3)(x)
outputs = layers.Dense(units=1, activation="sigmoid")(x)
# 定义模型。
model = keras.Model(inputs, outputs, name="3dcnn")
return model
# 构建模型。
model = get_model(width=128, height=128, depth=64)
model.summary()
Model: "3dcnn"
┏━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━┳━━━━━━━━━━━━━━━━━━━━━━━━━━━┳━━━━━━━━━━━━┓ ┃ Layer (type) ┃ Output Shape ┃ Param # ┃ ┡━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━╇━━━━━━━━━━━━━━━━━━━━━━━━━━━╇━━━━━━━━━━━━┩ │ input_layer (InputLayer) │ (None, 128, 128, 64, 1) │ 0 │ ├─────────────────────────────────┼───────────────────────────┼────────────┤ │ conv3d (Conv3D) │ (None, 126, 126, 62, 64) │ 1,792 │ ├─────────────────────────────────┼───────────────────────────┼────────────┤ │ max_pooling3d (MaxPooling3D) │ (无, 63, 63, 31, 64) │ 0 │ ├─────────────────────────────────┼───────────────────────────┼────────────┤ │ batch_normalization │ (无, 63, 63, 31, 64) │ 256 │ │ (BatchNormalization) │ │ │ ├─────────────────────────────────┼───────────────────────────┼────────────┤ │ conv3d_1 (Conv3D) │ (无, 61, 61, 29, 64) │ 110,656 │ ├─────────────────────────────────┼───────────────────────────┼────────────┤ │ max_pooling3d_1 (MaxPooling3D) │ (无, 30, 30, 14, 64) │ 0 │ ├─────────────────────────────────┼───────────────────────────┼────────────┤ │ batch_normalization_1 │ (无, 30, 30, 14, 64) │ 256 │ │ (BatchNormalization) │ │ │ ├─────────────────────────────────┼───────────────────────────┼────────────┤ │ conv3d_2 (Conv3D) │ (无, 28, 28, 12, 128) │ 221,312 │ ├─────────────────────────────────┼───────────────────────────┼────────────┤ │ max_pooling3d_2 (MaxPooling3D) │ (无, 14, 14, 6, 128) │ 0 │ ├─────────────────────────────────┼───────────────────────────┼────────────┤ │ batch_normalization_2 │ (无, 14, 14, 6, 128) │ 512 │ │ (批量归一化) │ │ │ ├─────────────────────────────────┼───────────────────────────┼────────────┤ │ conv3d_3 (卷积3D) │ (无, 12, 12, 4, 256) │ 884,992 │ ├─────────────────────────────────┼───────────────────────────┼────────────┤ │ max_pooling3d_3 (最大池化3D) │ (无, 6, 6, 2, 256) │ 0 │ ├─────────────────────────────────┼───────────────────────────┼────────────┤ │ batch_normalization_3 │ (无, 6, 6, 2, 256) │ 1,024 │ │ (批量归一化) │ │ │ ├─────────────────────────────────┼───────────────────────────┼────────────┤ │ global_average_pooling3d │ (无, 256) │ 0 │ │ (全局平均池化3D) │ │ │ ├─────────────────────────────────┼───────────────────────────┼────────────┤ │ dense (全连接层) │ (无, 512) │ 131,584 │ ├─────────────────────────────────┼───────────────────────────┼────────────┤ │ dropout (丢弃) │ (无, 512) │ 0 │ ├─────────────────────────────────┼───────────────────────────┼────────────┤ │ dense_1 (全连接层) │ (无, 1) │ 513 │ └─────────────────────────────────┴───────────────────────────┴────────────┘
总参数量: 1,352,897 (5.16 MB)
可训练参数: 1,351,873 (5.16 MB)
非可训练参数: 1,024 (4.00 KB)
训练模型
# 编译模型。
initial_learning_rate = 0.0001
lr_schedule = keras.optimizers.schedules.ExponentialDecay(
initial_learning_rate, decay_steps=100000, decay_rate=0.96, staircase=True
)
model.compile(
loss="binary_crossentropy",
optimizer=keras.optimizers.Adam(learning_rate=lr_schedule),
metrics=["acc"],
run_eagerly=True,
)
# 定义回调函数。
checkpoint_cb = keras.callbacks.ModelCheckpoint(
"3d_image_classification.keras", save_best_only=True
)
early_stopping_cb = keras.callbacks.EarlyStopping(monitor="val_acc", patience=15)
# 训练模型,在每个周期结束时进行验证
epochs = 100
model.fit(
train_dataset,
validation_data=validation_dataset,
epochs=epochs,
shuffle=True,
verbose=2,
callbacks=[checkpoint_cb, early_stopping_cb],
)
第 1 轮/100
70/70 - 40s - 568ms/步 - acc: 0.5786 - loss: 0.7128 - val_acc: 0.5000 - val_loss: 0.8744
第 2 轮/100
70/70 - 26s - 370ms/步 - acc: 0.6000 - loss: 0.6760 - val_acc: 0.5000 - val_loss: 1.2741
第 3 轮/100
70/70 - 26s - 373ms/步 - acc: 0.5643 - loss: 0.6768 - val_acc: 0.5000 - val_loss: 1.4767
第 4 轮/100
70/70 - 26s - 376ms/步 - acc: 0.6643 - loss: 0.6671 - val_acc: 0.5000 - val_loss: 1.2609
第 5 轮/100
70/70 - 26s - 374ms/步 - acc: 0.6714 - loss: 0.6274 - val_acc: 0.5667 - val_loss: 0.6470
第 6 轮/100
70/70 - 26s - 372ms/步 - acc: 0.5929 - loss: 0.6492 - val_acc: 0.6667 - val_loss: 0.6022
第 7 轮/100
70/70 - 26s - 374ms/步 - acc: 0.5929 - loss: 0.6601 - val_acc: 0.5667 - val_loss: 0.6788
第 8 轮/100
70/70 - 26s - 378ms/步 - acc: 0.6000 - loss: 0.6559 - val_acc: 0.6667 - val_loss: 0.6090
第 9 轮/100
70/70 - 26s - 373ms/步 - acc: 0.6357 - loss: 0.6423 - val_acc: 0.6000 - val_loss: 0.6535
第 10 轮/100
70/70 - 26s - 374ms/步 - acc: 0.6500 - loss: 0.6127 - val_acc: 0.6500 - val_loss: 0.6204
第 11 轮/100
70/70 - 26s - 374ms/步 - acc: 0.6714 - loss: 0.5994 - val_acc: 0.7000 - val_loss: 0.6218
第 12 轮/100
70/70 - 26s - 374ms/步 - acc: 0.6714 - loss: 0.5980 - val_acc: 0.7167 - val_loss: 0.5069
第 13 轮/100
70/70 - 26s - 369ms/步 - acc: 0.7214 - loss: 0.6003 - val_acc: 0.7833 - val_loss: 0.5182
第 14 轮/100
70/70 - 26s - 372ms/步 - acc: 0.6643 - loss: 0.6076 - val_acc: 0.7167 - val_loss: 0.5613
第 15 轮/100
70/70 - 26s - 373ms/步 - acc: 0.6571 - loss: 0.6359 - val_acc: 0.6167 - val_loss: 0.6184
第 16 轮/100
70/70 - 26s - 374ms/步 - acc: 0.6429 - loss: 0.6053 - val_acc: 0.7167 - val_loss: 0.5258
第 17 轮/100
70/70 - 26s - 370ms/步 - acc: 0.6786 - loss: 0.6119 - val_acc: 0.5667 - val_loss: 0.8481
第 18 轮/100
70/70 - 26s - 372ms/步 - acc: 0.6286 - loss: 0.6298 - val_acc: 0.6667 - val_loss: 0.5709
第 19 轮/100
70/70 - 26s - 372ms/步 - acc: 0.7214 - loss: 0.5979 - val_acc: 0.5833 - val_loss: 0.6730
第 20 轮/100
70/70 - 26s - 372ms/步 - acc: 0.7571 - loss: 0.5224 - val_acc: 0.7167 - val_loss: 0.5710
第 21 轮/100
70/70 - 26s - 372ms/步 - acc: 0.7357 - loss: 0.5606 - val_acc: 0.7167 - val_loss: 0.5444
第 22 轮/100
70/70 - 26s - 372ms/步 - acc: 0.7357 - loss: 0.5334 - val_acc: 0.5667 - val_loss: 0.7919
第 23 轮/100
70/70 - 26s - 373ms/步 - acc: 0.7071 - loss: 0.5337 - val_acc: 0.5167 - val_loss: 0.9527
第 24 轮/100
70/70 - 26s - 371ms/步 - acc: 0.7071 - loss: 0.5635 - val_acc: 0.7167 - val_loss: 0.5333
第 25 轮/100
70/70 - 26s - 373ms/步 - acc: 0.7643 - loss: 0.4787 - val_acc: 0.6333 - val_loss: 1.0172
第 26 轮/100
70/70 - 26s - 372ms/步 - acc: 0.7357 - loss: 0.5535 - val_acc: 0.6500 - val_loss: 0.6926
第 27 轮/100
70/70 - 26s - 370ms/步 - acc: 0.7286 - loss: 0.5608 - val_acc: 0.5000 - val_loss: 3.3032
第 28 轮/100
70/70 - 26s - 370ms/步 - acc: 0.7429 - loss: 0.5436 - val_acc: 0.6500 - val_loss: 0.6438
<keras.src.callbacks.history.History at 0x7fc5b923e810>
重要的是要注意样本数量非常少(仅 200),并且我们没有指定随机种子。因此,您可以预期结果的显著变化。完整的数据集由超过 1000 个 CT 扫描构成,您可以在 这里 找到。使用完整数据集时,达到了 83% 的准确率。在这两种情况下,分类性能的变化为 6-7%。
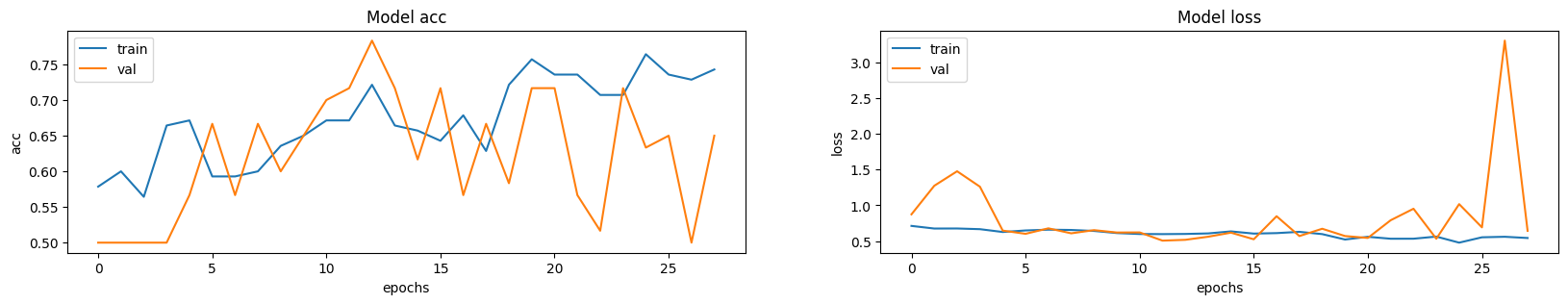
可视化模型性能
这里绘制了训练集和验证集的模型准确率和损失。由于验证集是类别平衡的,准确率提供了模型性能的无偏表示。
fig, ax = plt.subplots(1, 2, figsize=(20, 3))
ax = ax.ravel()
for i, metric in enumerate(["acc", "loss"]):
ax[i].plot(model.history.history[metric])
ax[i].plot(model.history.history["val_" + metric])
ax[i].set_title("Model {}".format(metric))
ax[i].set_xlabel("epochs")
ax[i].set_ylabel(metric)
ax[i].legend(["train", "val"])
对单个CT扫描进行预测
# 加载最佳权重。
model.load_weights("3d_image_classification.keras")
prediction = model.predict(np.expand_dims(x_val[0], axis=0))[0]
scores = [1 - prediction[0], prediction[0]]
class_names = ["normal", "abnormal"]
for score, name in zip(scores, class_names):
print(
"该模型对CT扫描是%s的自信度为%.2f percent"
% ((100 * score), name)
)
1/1 ━━━━━━━━━━━━━━━━━━━━ 0s 478ms/step
1/1 ━━━━━━━━━━━━━━━━━━━━ 0s 479ms/step
该模型对CT扫描是正常的自信度为32.99 percent
该模型对CT扫描是异常的自信度为67.01 percent
