DiscreteAliasUrn#
- class scipy.stats.sampling.DiscreteAliasUrn(dist, *, domain=None, urn_factor=1, random_state=None)#
离散别名-骨灰盒方法。
此方法用于从具有有限域的单变量离散分布中采样。它使用大小为 \(N\) 的概率向量或具有有限支持的概率质量函数来生成来自该分布的随机数。
- 参数:
- dist类数组或对象,可选
分布的概率向量(PV)。如果PV不可用,则期望一个具有
pmf方法的类实例。PMF的签名期望为:def pmf(self, k: int) -> float。即它应接受一个Python整数并返回一个Python浮点数。- 领域int, 可选
支持PMF。如果概率向量 (
pv) 不可用,则必须给定一个有限域。即PMF必须有一个有限的支持。默认是None。当None时:如果分布对象 dist 提供了
support方法,它将用于设置分布的域。否则,支持被假定为
(0, len(pv))。当此参数与概率向量一起传递时,domain[0]用于将分布从(0, len(pv))重新定位到(domain[0], domain[0]+len(pv)),而domain[1]被忽略。有关更详细的解释,请参见注释和教程。
- urn_factorfloat, 可选
骨灰瓮表的大小 相对于 概率向量的大小。它不能小于1。较大的表会导致生成时间更快,但需要更昂贵的设置。默认值为1。
- random_state{None, int,}
用于生成均匀随机数流的底层 NumPy 随机数生成器的 NumPy 随机数生成器或种子。如果 random_state 是 None(或 np.random),则使用
numpy.random.RandomState单例。如果 random_state 是整数,则使用一个新的RandomState实例,并以 random_state 为种子。如果 random_state 已经是Generator或RandomState实例,则使用该实例。
方法
rvs([size, random_state])来自发行版的示例。
set_random_state([random_state])设置底层均匀随机数生成器。
注释
当有限概率向量可用或分布的概率质量函数(PMF)可用时,此方法有效。如果仅提供PMF,则还必须给出PMF的*有限*支持(域)。建议首先通过在支持的每个点上评估PMF来获得概率向量,然后使用它。
如果给定一个概率向量,它必须是一个一维的非负浮点数数组,且不包含任何
inf或nan值。此外,必须至少有一个非零项,否则会引发异常。默认情况下,概率向量从0开始索引。然而,这可以通过传递一个
domain参数来改变。当domain与PV一起给出时,它会将分布从(0, len(pv))重新定位到(domain[0],domain[0] + len(pv))。在这种情况下,domain[1]被忽略。参数
urn_factor可以增加以加快生成速度,但会增加设置时间。此方法使用一个表来进行随机变量生成。urn_factor控制此表相对于概率向量(或支持的宽度,在没有PV的情况下)的大小。由于此表在设置时计算,增加此参数会线性增加所需的设置时间。建议将此参数保持在2以下。参考文献
[1]UNU.RAN 参考手册, 第5.8.2节, “DAU - (离散) 别名-Urn方法”, http://statmath.wu.ac.at/software/unuran/doc/unuran.html#DAU
[2]A.J. Walker (1977). 一种生成具有一般分布的离散随机变量的有效方法,ACM Trans. Math. Software 3, pp. 253-256。
示例
>>> from scipy.stats.sampling import DiscreteAliasUrn >>> import numpy as np
要使用概率向量创建随机数生成器,请使用:
>>> pv = [0.1, 0.3, 0.6] >>> urng = np.random.default_rng() >>> rng = DiscreteAliasUrn(pv, random_state=urng)
RNG 已经设置好。现在,我们可以使用 rvs 方法从分布中生成样本:
>>> rvs = rng.rvs(size=1000)
为了验证随机变量是否遵循给定的分布,我们可以使用卡方检验(作为拟合优度的度量):
>>> from scipy.stats import chisquare >>> _, freqs = np.unique(rvs, return_counts=True) >>> freqs = freqs / np.sum(freqs) >>> freqs array([0.092, 0.292, 0.616]) >>> chisquare(freqs, pv).pvalue 0.9993602047563164
由于p值非常高,我们未能拒绝原假设,即观察到的频率与预期频率相同。因此,我们可以安全地假设变量是从给定的分布中生成的。请注意,这仅证明了算法的正确性,而不是样本的质量。
如果无法提供 PV,也可以传递一个具有 PMF 方法和有限域的类实例。
>>> urng = np.random.default_rng() >>> class Binomial: ... def __init__(self, n, p): ... self.n = n ... self.p = p ... def pmf(self, x): ... # note that the pmf doesn't need to be normalized. ... return self.p**x * (1-self.p)**(self.n-x) ... def support(self): ... return (0, self.n) ... >>> n, p = 10, 0.2 >>> dist = Binomial(n, p) >>> rng = DiscreteAliasUrn(dist, random_state=urng)
现在,我们可以使用 rvs 方法从分布中采样,并测量样本的拟合优度:
>>> rvs = rng.rvs(1000) >>> _, freqs = np.unique(rvs, return_counts=True) >>> freqs = freqs / np.sum(freqs) >>> obs_freqs = np.zeros(11) # some frequencies may be zero. >>> obs_freqs[:freqs.size] = freqs >>> pv = [dist.pmf(i) for i in range(0, 11)] >>> pv = np.asarray(pv) / np.sum(pv) >>> chisquare(obs_freqs, pv).pvalue 0.9999999999999999
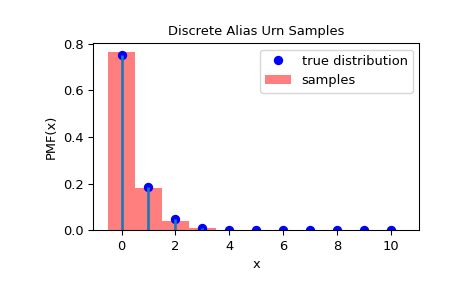
为了检查样本是否从正确的分布中抽取,我们可以可视化样本的直方图:
>>> import matplotlib.pyplot as plt >>> rvs = rng.rvs(1000) >>> fig = plt.figure() >>> ax = fig.add_subplot(111) >>> x = np.arange(0, n+1) >>> fx = dist.pmf(x) >>> fx = fx / fx.sum() >>> ax.plot(x, fx, 'bo', label='true distribution') >>> ax.vlines(x, 0, fx, lw=2) >>> ax.hist(rvs, bins=np.r_[x, n+1]-0.5, density=True, alpha=0.5, ... color='r', label='samples') >>> ax.set_xlabel('x') >>> ax.set_ylabel('PMF(x)') >>> ax.set_title('Discrete Alias Urn Samples') >>> plt.legend() >>> plt.show()
要设置
urn_factor,请使用:>>> rng = DiscreteAliasUrn(pv, urn_factor=2, random_state=urng)
这使用了一个两倍于概率向量大小的表格来从分布中生成随机变量。