dendrogram#
- scipy.cluster.hierarchy.dendrogram(Z, p=30, truncate_mode=None, color_threshold=None, get_leaves=True, orientation='top', labels=None, count_sort=False, distance_sort=False, show_leaf_counts=True, no_plot=False, no_labels=False, leaf_font_size=None, leaf_rotation=None, leaf_label_func=None, show_contracted=False, link_color_func=None, ax=None, above_threshold_color='C0')[源代码][源代码]#
将层次聚类绘制为树状图。
树状图通过绘制一个U形链接来展示每个集群的组成,该链接连接一个非单一集群及其子集群。U形链接的顶部表示集群合并。U形链接的两条腿表示合并的集群。U形链接两条腿的长度表示子集群之间的距离。这也是两个子集群中原始观测值之间的共表距离。
- 参数:
- Zndarray
链接矩阵编码了要渲染为树状图的层次聚类。有关
Z格式的更多信息,请参见linkage函数。- pint, 可选
truncate_mode的p参数。- truncate_modestr, 可选
当从其派生出链接的原始观测矩阵较大时,树状图可能难以阅读。截断用于压缩树状图。有几种模式:
None不执行截断(默认)。注意:
'none'是None的别名,保留以保持向后兼容性。'lastp'在链接中形成的最后一个
p非单例簇是链接中唯一的非叶节点;它们对应于Z中的行Z[n-p-2:end]。所有其他非单例簇都被收缩为叶节点。'level'显示的树状图树不超过
p层。一个“层”包括从最终合并开始的所有节点,这些节点距离最终合并有p次合并。注意:
'mtica'是'level'的别名,保留以实现向后兼容。
- color_threshold双精度,可选
为简洁起见,设 \(t\) 为
color_threshold。如果 \(k\) 是切割阈值 \(t\) 下的第一个节点,则将簇节点 \(k\) 下方的所有后代链接涂上相同的颜色。所有连接距离大于或等于阈值的节点的链接都用默认的 matplotlib 颜色'C0'着色。如果 \(t\) 小于或等于零,则所有节点都涂上'C0'。如果color_threshold为 None 或 ‘default’,对应于 MATLAB(TM) 行为,阈值设置为0.7*max(Z[:,2])。- get_leavesbool, 可选
在结果字典中包含一个列表
R['leaves']=H。对于每个 \(i\),H[i] == j,聚类节点j出现在从左到右遍历叶子的位置i中,其中 \(j < 2n-1\) 且 \(i < n\)。- 方向str, 可选
绘制树状图的方向,可以是以下字符串中的任何一个:
'top'将根节点置于顶部,并向下绘制后代链接。(默认)。
'bottom'将根节点绘制在底部,并向上绘制后代链接。
'left'将根节点绘制在左侧,并将后代链接绘制在右侧。
'right'将根节点绘制在右侧,并将后代链接绘制在左侧。
- 标签ndarray,可选
默认情况下,
labels是 None,因此使用原始观测的索引来标记叶节点。否则,这是一个大小为 \(n\) 的序列,其中n == Z.shape[0] + 1。如果labels[i]值对应于原始观测而不是非单例聚类,则labels[i]值是放在第 \(i\) 个叶节点下的文本。- 计数排序str 或 bool, 可选
对于每个节点 n,其两个后代链接的绘制顺序(从左到右)由该参数决定,该参数可以是以下值之一:
False什么都没有做。
'ascending'或True在其簇中拥有最少原始对象的子节点首先被绘制。
'descending'在其簇中拥有最多原始对象的子节点首先被绘制。
注意,
distance_sort和count_sort不能同时为 True。- distance_sortstr 或 bool, 可选
对于每个节点 n,其两个后代链接的绘制顺序(从左到右)由该参数决定,该参数可以是以下值之一:
False什么都没有做。
'ascending'或True首先绘制其直接后代之间距离最小的子节点。
'descending'具有直接后代之间最大距离的子节点首先被绘制。
注意
distance_sort和count_sort不能同时为 True。- show_leaf_countsbool, 可选
当为 True 时,表示 \(k>1\) 个原始观测值的叶节点会用括号标注它们包含的观测值数量。
- no_plotbool, 可选
当设置为 True 时,最终的渲染不会执行。这在只需要用于渲染的数据结构,或者 matplotlib 不可用时非常有用。
- 无标签bool, 可选
当为 True 时,在树状图的渲染中,叶节点旁边不会出现标签。
- leaf_rotation双精度,可选
指定旋转叶标签的角度(以度为单位)。未指定时,旋转角度基于树状图中的节点数量(默认值为0)。
- leaf_font_sizeint, 可选
指定叶标签的字体大小(以点为单位)。未指定时,大小基于树状图中节点的数量。
- leaf_label_funclambda 或 函数,可选
当
leaf_label_func是一个可调用函数时,对于每个簇索引为 \(k < 2n-1\) 的叶子节点,该函数应返回一个包含叶子标签的字符串。索引 \(k < n\) 对应于原始观测值,而索引 \(k \geq n\) 对应于非单例簇。
例如,要标记单例节点及其节点ID,非单例节点及其ID、计数和不一致系数,只需执行:
# First define the leaf label function. def llf(id): if id < n: return str(id) else: return '[%d %d %1.2f]' % (id, count, R[n-id,3]) # The text for the leaf nodes is going to be big so force # a rotation of 90 degrees. dendrogram(Z, leaf_label_func=llf, leaf_rotation=90) # leaf_label_func can also be used together with ``truncate_mode``, # in which case you will get your leaves labeled after truncation: dendrogram(Z, leaf_label_func=llf, leaf_rotation=90, truncate_mode='level', p=2)
- show_contractedbool, 可选
当为True时,非单例节点收缩到叶节点的高度将沿着连接该叶节点的链接绘制为十字。这实际上仅在使用了截断时才有用(参见
truncate_mode参数)。- link_color_func可调用,可选
如果提供了 link_color_function,它将被调用,每个非单例的 id 对应于每个 U 形链接,它将绘制这些链接。该函数应返回要绘制的链接的颜色,编码为 matplotlib 颜色字符串代码。例如:
dendrogram(Z, link_color_func=lambda k: colors[k])
使用
colors[k]为每个未截断的非单例节点k下的直接链接着色。- axmatplotlib Axes 实例,可选
如果为 None 且 no_plot 不为 True,树状图将绘制在当前坐标轴上。否则,如果 no_plot 不为 True,树状图将绘制在给定的
Axes实例上。这在树状图是更复杂图形的一部分时非常有用。- above_threshold_colorstr, 可选
这个 matplotlib 颜色字符串设置了颜色阈值上方链接的颜色。默认值是
'C0'。
- 返回:
- Rdict
一个计算用于渲染树状图的数据结构字典。它具有以下键:
'color_list'颜色名称列表。第 k 个元素表示第 k 个链接的颜色。
'icoord'和'dcoord'每个都是列表的列表。设
icoord = [I1, I2, ..., Ip]其中Ik = [xk1, xk2, xk3, xk4]和dcoord = [D1, D2, ..., Dp]其中Dk = [yk1, yk2, yk3, yk4],那么绘制的第 k 个链接是(xk1, yk1)-(xk2, yk2)-(xk3, yk3)-(xk4, yk4)。'ivl'对应于叶节点的标签列表。
'leaves'对于每个 i,
H[i] == j,聚类节点j出现在从左到右遍历叶子的位置i中,其中 \(j < 2n-1\) 且 \(i < n\)。如果j小于n,则第i个叶子节点对应于一个原始观测值。否则,它对应于一个非单例聚类。'leaves_color_list'颜色名称列表。第 k 个元素表示第 k 个叶子的颜色。
注释
期望
Z[:,2]中的距离是单调的,否则在树状图中会出现交叉。示例
>>> import numpy as np >>> from scipy.cluster import hierarchy >>> import matplotlib.pyplot as plt
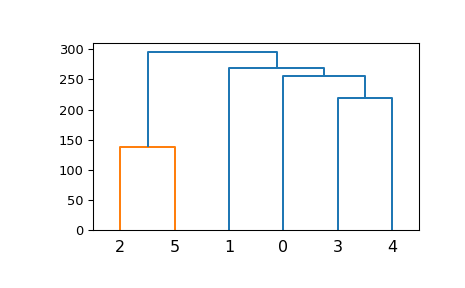
一个非常基础的例子:
>>> ytdist = np.array([662., 877., 255., 412., 996., 295., 468., 268., ... 400., 754., 564., 138., 219., 869., 669.]) >>> Z = hierarchy.linkage(ytdist, 'single') >>> plt.figure() >>> dn = hierarchy.dendrogram(Z)
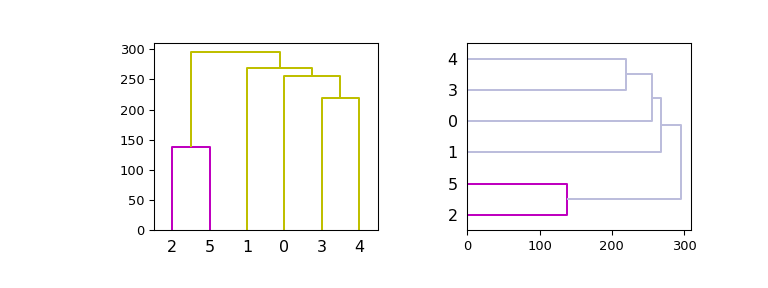
现在,在给定的轴上绘图,改进配色方案并使用垂直和水平方向:
>>> hierarchy.set_link_color_palette(['m', 'c', 'y', 'k']) >>> fig, axes = plt.subplots(1, 2, figsize=(8, 3)) >>> dn1 = hierarchy.dendrogram(Z, ax=axes[0], above_threshold_color='y', ... orientation='top') >>> dn2 = hierarchy.dendrogram(Z, ax=axes[1], ... above_threshold_color='#bcbddc', ... orientation='right') >>> hierarchy.set_link_color_palette(None) # reset to default after use >>> plt.show()