logrank#
- scipy.stats.logrank(x, y, alternative='two-sided')[源代码][源代码]#
通过对数秩检验比较两个样本的生存分布。
- 参数:
- x, y类似数组或CensoredData
基于经验生存函数进行比较的样本。
- 替代方案{‘双侧’, ‘小于’, ‘大于’}, 可选
定义备择假设。
零假设是两组(例如 X 和 Y)的生存分布是相同的。
以下备选假设 [4] 可用(默认是 ‘双侧’):
‘双侧’: 两组的生存分布不相同。
‘less’: 群体 X 的生存受到青睐:在某些时候,群体 X 的失效率函数低于群体 Y 的失效率函数。
‘greater’: 群体 Y 的生存被偏好: 群体 X 的失效率函数在某些时刻大于群体 Y 的失效率函数。
- 返回:
- resLogRankResult
一个包含属性的对象:
- 统计浮点数 ndarray
计算的统计量(定义如下)。其大小是大多数其他对数秩检验实现返回的大小平方根。
- p值浮点数 ndarray
测试的计算p值。
注释
logrank 检验 [1] 比较了在零假设下,两个样本来自同一分布时,观察到的事件数与预期的事件数。该统计量是
\[Z_i = \frac{\sum_{j=1}^J(O_{i,j}-E_{i,j})}{\sqrt{\sum_{j=1}^J V_{i,j}}} \rightarrow \mathcal{N}(0,1)\]哪里
\[E_{i,j} = O_j \frac{N_{i,j}}{N_j}, \qquad V_{i,j} = E_{i,j} \left(\frac{N_j-O_j}{N_j}\right) \left(\frac{N_j-N_{i,j}}{N_j-1}\right),\]\(i\) 表示组(即它可以取值 \(x\) 或 \(y\),或者它可以被省略以指代合并样本),\(j\) 表示时间(事件发生的时间),\(N\) 是在事件发生前处于风险中的受试者数量,\(O\) 是在该时间点观察到的事件数量。
statistic\(Z_x\) 由logrank返回的是许多其他实现返回的统计量的(带符号的)平方根。在零假设下,\(Z_x**2\) 渐近地服从自由度为1的卡方分布。因此,\(Z_x\) 渐近地服从标准正态分布。使用 \(Z_x\) 的优点是保留了符号信息(即观察到的事件数量是否倾向于少于或大于零假设下的预期数量),从而使scipy.stats.logrank能够提供单侧备择假设。参考文献
[1]Mantel N. “生存数据的评估及其考虑中产生的两个新的秩次统计量。” 《癌症化疗报告》,50(3):163-170,PMID: 5910392,1966
[2]Bland, Altman, “The logrank test”, BMJ, 328:1073, DOI:10.1136/bmj.328.7447.1073, 2004
[3]“Logrank 检验”,维基百科,https://en.wikipedia.org/wiki/Logrank_test
[4]Brown, Mark. “对数秩检验中方差的选取。” 《生物计量学》71.1 (1984): 65-74.
[5]Klein, John P., 和 Melvin L. Moeschberger. 生存分析:处理截尾和截断数据的技术. 第1230卷. 纽约: Springer, 2003.
示例
参考文献 [2] 比较了两类复发性恶性胶质瘤患者的生存时间。以下样本记录了每位患者参与研究的时间(周数)。使用
scipy.stats.CensoredData类是因为数据是右删失的:未删失的观测对应于观察到的死亡,而删失的观测对应于患者因其他原因离开研究。>>> from scipy import stats >>> x = stats.CensoredData( ... uncensored=[6, 13, 21, 30, 37, 38, 49, 50, ... 63, 79, 86, 98, 202, 219], ... right=[31, 47, 80, 82, 82, 149] ... ) >>> y = stats.CensoredData( ... uncensored=[10, 10, 12, 13, 14, 15, 16, 17, 18, 20, 24, 24, ... 25, 28,30, 33, 35, 37, 40, 40, 46, 48, 76, 81, ... 82, 91, 112, 181], ... right=[34, 40, 70] ... )
我们可以如下计算并可视化两组的经验生存函数。
>>> import numpy as np >>> import matplotlib.pyplot as plt >>> ax = plt.subplot() >>> ecdf_x = stats.ecdf(x) >>> ecdf_x.sf.plot(ax, label='Astrocytoma') >>> ecdf_y = stats.ecdf(y) >>> ecdf_y.sf.plot(ax, label='Glioblastoma') >>> ax.set_xlabel('Time to death (weeks)') >>> ax.set_ylabel('Empirical SF') >>> plt.legend() >>> plt.show()
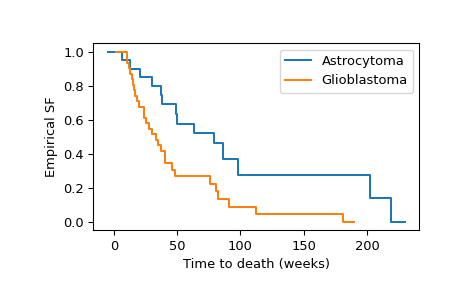
对经验生存函数的视觉检查表明,两组之间的生存时间往往不同。为了正式评估这种差异在1%水平上是否显著,我们使用对数秩检验。
>>> res = stats.logrank(x=x, y=y) >>> res.statistic -2.73799 >>> res.pvalue 0.00618
p 值小于 1%,因此我们可以认为数据是支持备择假设的证据,即两个生存函数之间存在差异。