肯德尔τ#
- scipy.stats.kendalltau(x, y, *, nan_policy='propagate', method='auto', variant='b', alternative='two-sided')[源代码][源代码]#
计算 Kendall’s tau,一种用于有序数据的关联度量。
肯德尔的 tau 是衡量两个排名之间对应关系的一个指标。接近 1 的值表示强烈的一致,接近 -1 的值表示强烈的分歧。这里实现了肯德尔 tau 的两种变体:tau-b(默认)和 tau-c(也称为斯图尔特的 tau-c)。它们仅在如何归一化到 -1 到 1 的范围上有所不同;假设检验(它们的 p 值)是相同的。肯德尔原始的 tau-a 没有单独实现,因为在没有平局的情况下,tau-b 和 tau-c 都简化为 tau-a。
- 参数:
- x, yarray_like
排名的数组,形状相同。如果数组不是一维的,它们将被展平为一维。
- nan_policy{‘propagate’, ‘raise’, ‘omit’}, 可选
定义当输入包含 nan 时的处理方式。以下选项可用(默认是 ‘propagate’):
‘propagate’: 返回 nan
‘raise’: 抛出一个错误
‘omit’: 执行计算时忽略 nan 值
- 方法{‘auto’, ‘asymptotic’, ‘exact’}, 可选
定义用于计算p值的方法 [5] 。以下选项可用(默认是’auto’):
‘auto’: 根据速度和准确性之间的权衡选择适当的方法
‘asymptotic’: 使用适用于大样本的有效正态近似
‘exact’: 计算精确的 p 值,但只能在不存在平局的情况下使用。随着样本量的增加,’exact’ 计算时间可能会增长,结果可能会失去一些精度。
- 变体{‘b’, ‘c’}, 可选
定义返回的 Kendall’s tau 的变体。默认是 ‘b’。
- 替代方案{‘双侧’, ‘小于’, ‘大于’}, 可选
定义备择假设。默认是’双侧’。以下选项可用:
‘双侧’: 秩相关性非零
‘less’: 排名相关性为负(小于零)
‘greater’: 秩相关是正的(大于零)
- 返回:
- res重要性结果
一个包含属性的对象:
- 统计浮动
tau统计量。
- p值浮动
对于零假设为无关联的假设检验,p值为,tau = 0。
参见
spearmanr计算斯皮尔曼等级相关系数。
theilslopes计算一组点 (x, y) 的 Theil-Sen 估计量。
weightedtau计算Kendall’s tau的加权版本。
注释
所使用的 Kendall’s tau 的定义是 [2]:
tau_b = (P - Q) / sqrt((P + Q + T) * (P + Q + U)) tau_c = 2 (P - Q) / (n**2 * (m - 1) / m)
其中 P 是和谐对的数量,Q 是不和谐对的数量,T 是仅在 x 中打结的数量,U 是仅在 y 中打结的数量。如果同一个对在 x 和 y 中都打结,则不计入 T 或 U。n 是样本总数,m 是 x 或 y 中较小的一个的唯一值的数量。
参考文献
[1]Maurice G. Kendall, “一种新的秩相关度量”, Biometrika 第30卷, 第1/2期, 第81-93页, 1938年。
[2]Maurice G. Kendall, “排名问题中的平局处理”, Biometrika 第33卷, 第3期, 第239-251页. 1945.
[3]Gottfried E. Noether, “非参数统计元素”, John Wiley & Sons, 1967.
[4]Peter M. Fenwick, “一种新的累积频率表数据结构”, 《软件:实践与经验》, 第24卷, 第3期, 第327-336页, 1994年。
[5]Maurice G. Kendall, 《秩相关方法》(第4版),Charles Griffin & Co., 1970年。
[6]Kershenobich, D., Fierro, F. J., & Rojkind, M. (1970). 游离脯氨酸池与人类肝硬化中胶原含量的关系。临床研究杂志, 49(12), 2246-2249.
[7]Hollander, M., Wolfe, D. A., & Chicken, E. (2013). 非参数统计方法. John Wiley & Sons.
[8]B. Phipson and G. K. Smyth. “Permutation P-values Should Never Be Zero: Calculating Exact P-values When Permutations Are Randomly Drawn.” Statistical Applications in Genetics and Molecular Biology 9.1 (2010).
示例
考虑来自 [6] 的数据,该研究探讨了游离脯氨酸(一种氨基酸)与总胶原蛋白(一种常在结缔组织中发现的蛋白质)在不健康人肝脏中的关系。
下面的
x和y数组记录了两种化合物的测量值。观察值是成对的:每次游离脯氨酸测量都取自与相同索引处的总胶原蛋白测量相同的肝脏。>>> import numpy as np >>> # total collagen (mg/g dry weight of liver) >>> x = np.array([7.1, 7.1, 7.2, 8.3, 9.4, 10.5, 11.4]) >>> # free proline (μ mole/g dry weight of liver) >>> y = np.array([2.8, 2.9, 2.8, 2.6, 3.5, 4.6, 5.0])
这些数据在 [7] 中使用斯皮尔曼相关系数进行了分析,该统计量类似于肯德尔τ,因为它也对样本之间的序数相关性敏感。让我们使用肯德尔τ进行类似的研究。
>>> from scipy import stats >>> res = stats.kendalltau(x, y) >>> res.statistic 0.5499999999999999
这个统计量的值对于具有强正序相关的样本往往会很高(接近1),对于具有强负序相关的样本会很低(接近-1),而对于具有弱序相关的样本,其值的大小会很小(接近零)。
测试是通过将统计量的观测值与零分布进行比较来进行的:零分布是在总胶原蛋白和游离脯氨酸测量相互独立的零假设下得出的统计量值的分布。
在这个测试中,对于没有并列的大样本,零分布近似为正态分布,其方差为
(2*(2*n + 5))/(9*n*(n - 1)),其中n = len(x)。>>> import matplotlib.pyplot as plt >>> n = len(x) # len(x) == len(y) >>> var = (2*(2*n + 5))/(9*n*(n - 1)) >>> dist = stats.norm(scale=np.sqrt(var)) >>> z_vals = np.linspace(-1.25, 1.25, 100) >>> pdf = dist.pdf(z_vals) >>> fig, ax = plt.subplots(figsize=(8, 5)) >>> def plot(ax): # we'll reuse this ... ax.plot(z_vals, pdf) ... ax.set_title("Kendall Tau Test Null Distribution") ... ax.set_xlabel("statistic") ... ax.set_ylabel("probability density") >>> plot(ax) >>> plt.show()
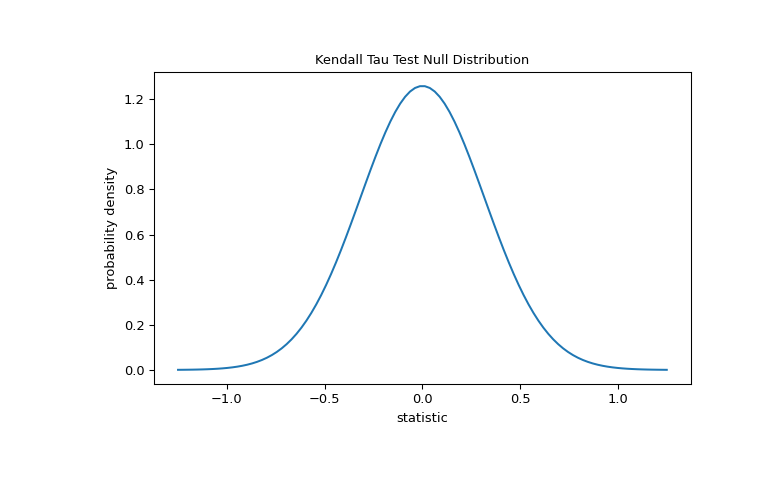
比较通过p值量化:零分布中与统计量的观测值同样极端或更极端的值的比例。在统计量为正的双侧检验中,零分布中大于转换后统计量的元素和零分布中小于观测统计量负值的元素都被视为“更极端”。
>>> fig, ax = plt.subplots(figsize=(8, 5)) >>> plot(ax) >>> pvalue = dist.cdf(-res.statistic) + dist.sf(res.statistic) >>> annotation = (f'p-value={pvalue:.4f}\n(shaded area)') >>> props = dict(facecolor='black', width=1, headwidth=5, headlength=8) >>> _ = ax.annotate(annotation, (0.65, 0.15), (0.8, 0.3), arrowprops=props) >>> i = z_vals >= res.statistic >>> ax.fill_between(z_vals[i], y1=0, y2=pdf[i], color='C0') >>> i = z_vals <= -res.statistic >>> ax.fill_between(z_vals[i], y1=0, y2=pdf[i], color='C0') >>> ax.set_xlim(-1.25, 1.25) >>> ax.set_ylim(0, 0.5) >>> plt.show()
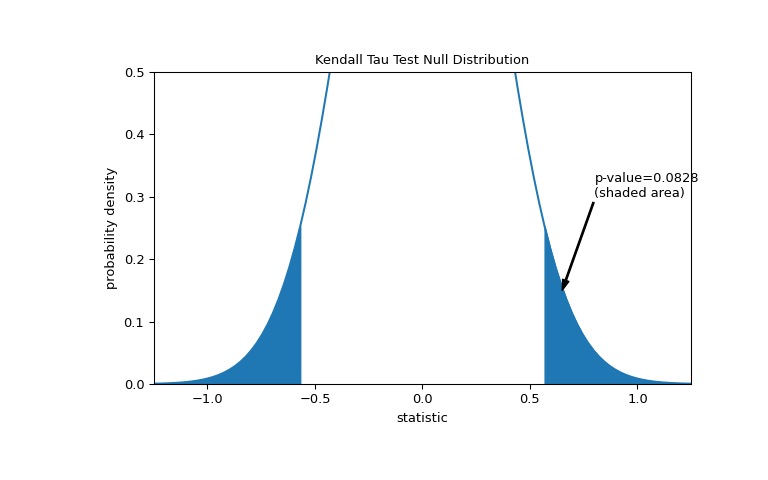
>>> res.pvalue 0.09108705741631495 # approximate p-value
请注意,曲线阴影区域与
kendalltau返回的 p 值之间存在轻微差异。这是因为我们的数据存在平局(ties),而我们忽略了kendalltau执行的零分布方差的平局校正。对于没有平局的样本,我们的图表阴影区域与kendalltau返回的 p 值将完全匹配。如果 p 值是“小”的——也就是说,如果从独立分布中抽取数据产生如此极端的统计值的概率很低——这可能被视为反对零假设而支持备择假设的证据:总胶原蛋白和游离脯氨酸的分布 不 是独立的。请注意:
反之则不然;也就是说,测试并不用于为零假设提供证据。
被视为“小”的值的阈值是一个在分析数据之前应做出的选择 [8] ,需考虑到假阳性(错误地拒绝零假设)和假阴性(未能拒绝错误的零假设)的风险。
小的p值并不是*大*效应的证据;相反,它们只能提供“显著”效应的证据,这意味着它们在零假设下不太可能发生。
对于没有中等大小关联的样本,
kendalltau可以精确计算 p 值。然而,在存在关联的情况下,kendalltau会采用渐近近似。尽管如此,我们可以使用排列检验来精确计算零分布:在零假设下,总胶原蛋白和游离脯氨酸是独立的,每个游离脯氨酸测量值与任何总胶原蛋白测量值的配对都是等可能的。因此,我们可以通过计算x和y之间每个可能元素配对下的统计量来形成一个 精确 的零分布。>>> def statistic(x): # explore all possible pairings by permuting `x` ... return stats.kendalltau(x, y).statistic # ignore pvalue >>> ref = stats.permutation_test((x,), statistic, ... permutation_type='pairings') >>> fig, ax = plt.subplots(figsize=(8, 5)) >>> plot(ax) >>> bins = np.linspace(-1.25, 1.25, 25) >>> ax.hist(ref.null_distribution, bins=bins, density=True) >>> ax.legend(['aymptotic approximation\n(many observations)', ... 'exact null distribution']) >>> plot(ax) >>> plt.show()
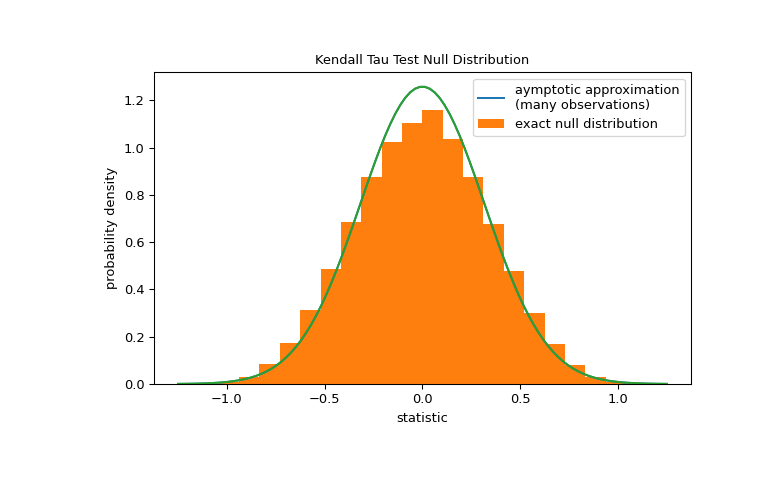
>>> ref.pvalue 0.12222222222222222 # exact p-value
注意,这里计算的确切p值与上面`kendalltau`返回的近似值之间存在显著分歧。对于有小样本且有结的情况,考虑进行置换检验以获得更准确的结果。