! [ -e /content ] && pip install -Uqq fastai # 在Colab上升级fastai要使用 fastai.medical.imaging,您需要:
conda install pyarrow
pip install pydicom kornia opencv-python scikit-image要在Google Colab上运行此教程,您需要取消注释以下两行并运行该单元:
#!conda 安装 pyarrow
#!pip 安装 pydicom kornia opencv-python scikit-image nbdevfrom fastai.basics import *
from fastai.callback.all import *
from fastai.vision.all import *
from fastai.medical.imaging import *
import pydicom
import pandas as pdfrom nbdev.showdoc import *胸部X光模型
在本教程中,我们将构建一个分类器,用于区分有气胸和没有气胸的胸部X光片。图像数据直接从DICOM源文件加载,因此不需要事先处理DICOM数据。本教程还将介绍DICOM图像的内容,并高层次地回顾如何评估分类器的结果。
下载和导入X射线DICOM文件
首先,我们将使用untar_data函数下载包含一部分(250个DICOM文件,约30MB)的_siim_small_文件夹,该文件夹来自于SIIM-ACR Pneumothorax Segmentation [1] 数据集。 下载的_siim_small_文件夹将被存储在您的_~/.fastai/data/_目录中。变量pneumothorax-source将存储_siim_small_文件夹的绝对路径,一旦下载完成。
pneumothorax_source = untar_data(URLs.SIIM_SMALL)siim_small 文件夹具有以下目录/文件结构:
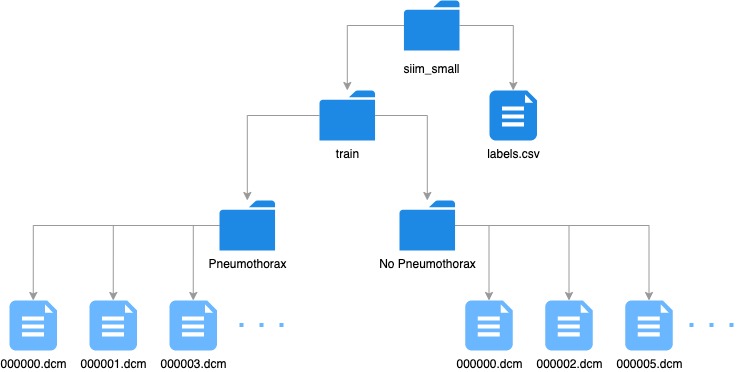
什么是DICOM?
DICOM(Digital Imaging and COmmunications in Medicine)是既定标准,规定了规则使得不同厂商的成像设备、计算机和医院之间能够交换医疗图像(X射线、MRI、CT)及相关信息。DICOM格式提供了一种合适的方式,以符合健康信息交换(HIE)标准,用于在设施之间传输健康相关数据,以及符合HL7标准,该标准是使临床应用能够交换数据的消息标准。
DICOM文件通常具有 .dcm 扩展名,并提供了一种以单独的“标签”存储数据的方法,例如患者信息和图像/像素数据。DICOM文件由一个头信息和图像数据集组成,这些信息被打包到一个文件中。通过提取这些标签中的数据,可以访问有关患者人口统计、研究参数等重要信息。
16位DICOM图像的值范围从 -32768 到 32768,而8位灰度图像的值范围从 0 到 255。DICOM图像中的值范围非常有用,因为它们与亨斯菲尔德标度相关,该标度是描述放射密度的定量标度。
绘制DICOM数据
为了分析我们的数据集,我们通过 get_dicom_files 函数加载 DICOM 文件的路径。在调用该函数时,我们将 train/ 附加到 pneumothorax_source 路径,以选择 DICOM 文件所在的文件夹。我们将每个 DICOM 文件的路径存储在 items 列表中。
items = get_dicom_files(pneumothorax_source/f"train/")接下来,我们使用 RandomSplitter 函数将 items 列表分割为训练 trn 和验证 val 列表:
trn,val = RandomSplitter()(items)Pydicom是一个用于解析DICOM文件的Python包,使得访问DICOM的header变得更简单,同时将原始的pixel_data转换为更易于操作的Python结构。fastai.medical.imaging使用pydicom.dcmread来加载DICOM文件。
为了绘制X光图像,我们可以在items列表中选择一个条目,并使用dcmread加载DICOM文件。
patient = 7
xray_sample = items[patient].dcmread()要查看header
xray_sampleDataset.file_meta -------------------------------
(0002, 0000) File Meta Information Group Length UL: 200
(0002, 0001) File Meta Information Version OB: b'\x00\x01'
(0002, 0002) Media Storage SOP Class UID UI: Secondary Capture Image Storage
(0002, 0003) Media Storage SOP Instance UID UI: 1.2.276.0.7230010.3.1.4.8323329.3297.1517875177.149805
(0002, 0010) Transfer Syntax UID UI: JPEG Baseline (Process 1)
(0002, 0012) Implementation Class UID UI: 1.2.276.0.7230010.3.0.3.6.0
(0002, 0013) Implementation Version Name SH: 'OFFIS_DCMTK_360'
-------------------------------------------------
(0008, 0005) Specific Character Set CS: 'ISO_IR 100'
(0008, 0016) SOP Class UID UI: Secondary Capture Image Storage
(0008, 0018) SOP Instance UID UI: 1.2.276.0.7230010.3.1.4.8323329.3297.1517875177.149805
(0008, 0020) Study Date DA: '19010101'
(0008, 0030) Study Time TM: '000000.00'
(0008, 0050) Accession Number SH: ''
(0008, 0060) Modality CS: 'CR'
(0008, 0064) Conversion Type CS: 'WSD'
(0008, 0090) Referring Physician's Name PN: ''
(0008, 103e) Series Description LO: 'view: PA'
(0010, 0010) Patient's Name PN: '6633c659-9249-443e-9851-b83782d1b111'
(0010, 0020) Patient ID LO: '6633c659-9249-443e-9851-b83782d1b111'
(0010, 0030) Patient's Birth Date DA: ''
(0010, 0040) Patient's Sex CS: 'M'
(0010, 1010) Patient's Age AS: '21'
(0018, 0015) Body Part Examined CS: 'CHEST'
(0018, 5101) View Position CS: 'PA'
(0020, 000d) Study Instance UID UI: 1.2.276.0.7230010.3.1.2.8323329.3297.1517875177.149804
(0020, 000e) Series Instance UID UI: 1.2.276.0.7230010.3.1.3.8323329.3297.1517875177.149803
(0020, 0010) Study ID SH: ''
(0020, 0011) Series Number IS: "1"
(0020, 0013) Instance Number IS: "1"
(0020, 0020) Patient Orientation CS: ''
(0028, 0002) Samples per Pixel US: 1
(0028, 0004) Photometric Interpretation CS: 'MONOCHROME2'
(0028, 0010) Rows US: 1024
(0028, 0011) Columns US: 1024
(0028, 0030) Pixel Spacing DS: [0.14300000000000002, 0.14300000000000002]
(0028, 0100) Bits Allocated US: 8
(0028, 0101) Bits Stored US: 8
(0028, 0102) High Bit US: 7
(0028, 0103) Pixel Representation US: 0
(0028, 2110) Lossy Image Compression CS: '01'
(0028, 2114) Lossy Image Compression Method CS: 'ISO_10918_1'
(7fe0, 0010) Pixel Data OB: Array of 161452 elements每个元素的解释超出了本教程的范围,但这个网站提供了有关每个条目的优质信息。
关于以上标签信息的一些关键要点:
- 像素数据 (7fe0 0010) - 这是存储原始像素数据的地方。每个图像平面中编码的像素顺序是从左到右,从上到下,即最左上角的像素(标记为1,1)是首先编码的。
- 光度解释 (0028, 0004) - 也称为色彩空间。在本例中为
单色2,其中像素数据表示为单一的单色图像平面,低值=黑暗,高值=明亮。如果色彩空间是单色,则低值=明亮,高值=黑暗。 - 每像素样本数 (0028, 0002) - 由于此图像是单色的,这应该是1。如果色彩空间是RGB,则这个值将是3。
- 存储位数 (0028 0101) - 每个像素样本存储的位数。典型的8位图像的像素范围在
0和255之间。 - 像素表示 (0028 0103) - 可以是无符号(0)或有符号(1)。
- 有损图像压缩 (0028 2110) -
00表示图像未经过有损压缩。01表示图像已经过有损压缩。 - 有损图像压缩方法 (0028 2114) - 指定使用的有损压缩类型(在此情况下,
ISO_10918_1表示JPEG有损压缩)。 - 像素数据 (7fe0, 0010) - 数组包含161452个元素,表示pydicom用于将像素数据转换为图像的图像像素数据。
PixelData 看起来是什么样的?
xray_sample.PixelData[:200]b'\xfe\xff\x00\xe0\x00\x00\x00\x00\xfe\xff\x00\xe0\x9cv\x02\x00\xff\xd8\xff\xdb\x00C\x00\x03\x02\x02\x02\x02\x02\x03\x02\x02\x02\x03\x03\x03\x03\x04\x06\x04\x04\x04\x04\x04\x08\x06\x06\x05\x06\t\x08\n\n\t\x08\t\t\n\x0c\x0f\x0c\n\x0b\x0e\x0b\t\t\n\x11\n\x0e\x0f\x10\x10\x11\x10\n\x0c\x12\x13\x12\x10\x13\x0f\x10\x10\x10\xff\xc0\x00\x0b\x08\x04\x00\x04\x00\x01\x01\x11\x00\xff\xc4\x00\x1d\x00\x00\x02\x03\x01\x01\x01\x01\x01\x00\x00\x00\x00\x00\x00\x00\x00\x04\x05\x02\x03\x06\x00\x01\x07\x08\t\xff\xc4\x00R\x10\x00\x02\x01\x03\x03\x02\x04\x03\x06\x05\x04\x00\x04\x01\x02\x17\x01\x02\x11\x00\x03!\x04\x121\x05A\x13"Qa\x06q\x81\x142\x91\xa1\xb1\xf0#B\xc1\xd1\xe1\x07\x15R\xf1\x16$3br\x08%4C&cs\x82\x92\xa2'由于解读 PixelData 的复杂性,pydicom 提供了一种简单的方法以方便形式获取它:pixel_array,该方法返回一个包含像素数据的 numpy.ndarray:
xray_sample.pixel_array, xray_sample.pixel_array.shape(array([[ 0, 0, 0, ..., 13, 13, 5],
[ 0, 0, 0, ..., 13, 13, 5],
[ 0, 0, 0, ..., 13, 12, 5],
...,
[ 0, 0, 0, ..., 5, 3, 0],
[ 0, 0, 0, ..., 6, 4, 0],
[ 0, 0, 0, ..., 8, 5, 0]], dtype=uint8),
(1024, 1024))您可以使用 show 函数来查看图像。
xray_sample.show()您还可以通过使用 from_dicoms 方便地创建一个数据框,其中包含数据集中所有图像的 tag 信息作为列。
dicom_dataframe = pd.DataFrame.from_dicoms(items)
dicom_dataframe[:5]| SpecificCharacterSet | SOPClassUID | SOPInstanceUID | StudyDate | StudyTime | AccessionNumber | Modality | ConversionType | ReferringPhysicianName | SeriesDescription | ... | LossyImageCompression | LossyImageCompressionMethod | fname | MultiPixelSpacing | PixelSpacing1 | img_min | img_max | img_mean | img_std | img_pct_window | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | ISO_IR 100 | 1.2.840.10008.5.1.4.1.1.7 | 1.2.276.0.7230010.3.1.4.8323329.6904.1517875201.850819 | 19010101 | 000000.00 | CR | WSD | view: PA | ... | 01 | ISO_10918_1 | C:\Users\tijme\.fastai\data\siim_small\train\No Pneumothorax\000000.dcm | 1 | 0.168 | 0 | 254 | 160.398039 | 53.854885 | 0.087029 | ||
| 1 | ISO_IR 100 | 1.2.840.10008.5.1.4.1.1.7 | 1.2.276.0.7230010.3.1.4.8323329.11028.1517875229.983789 | 19010101 | 000000.00 | CR | WSD | view: PA | ... | 01 | ISO_10918_1 | C:\Users\tijme\.fastai\data\siim_small\train\No Pneumothorax\000002.dcm | 1 | 0.143 | 0 | 250 | 114.524713 | 70.752315 | 0.326269 | ||
| 2 | ISO_IR 100 | 1.2.840.10008.5.1.4.1.1.7 | 1.2.276.0.7230010.3.1.4.8323329.11444.1517875232.977506 | 19010101 | 000000.00 | CR | WSD | view: PA | ... | 01 | ISO_10918_1 | C:\Users\tijme\.fastai\data\siim_small\train\No Pneumothorax\000005.dcm | 1 | 0.143 | 0 | 246 | 132.218334 | 73.023531 | 0.266901 | ||
| 3 | ISO_IR 100 | 1.2.840.10008.5.1.4.1.1.7 | 1.2.276.0.7230010.3.1.4.8323329.32219.1517875159.70802 | 19010101 | 000000.00 | CR | WSD | view: PA | ... | 01 | ISO_10918_1 | C:\Users\tijme\.fastai\data\siim_small\train\No Pneumothorax\000006.dcm | 1 | 0.171 | 0 | 255 | 153.405355 | 59.543063 | 0.144505 | ||
| 4 | ISO_IR 100 | 1.2.840.10008.5.1.4.1.1.7 | 1.2.276.0.7230010.3.1.4.8323329.32395.1517875160.396775 | 19010101 | 000000.00 | CR | WSD | view: PA | ... | 01 | ISO_10918_1 | C:\Users\tijme\.fastai\data\siim_small\train\No Pneumothorax\000007.dcm | 1 | 0.171 | 0 | 250 | 166.198407 | 50.008985 | 0.053009 |
5 rows × 42 columns
接下来,我们需要加载数据集的标签。我们使用pandas导入_labels.csv_文件并打印前五个条目。file列显示_.dcm_文件的相对路径,label列指示胸部X光片是否有气胸。
df = pd.read_csv(pneumothorax_source/f"labels.csv")
df.head()| file | label | |
|---|---|---|
| 0 | train/No Pneumothorax/000000.dcm | No Pneumothorax |
| 1 | train/Pneumothorax/000001.dcm | Pneumothorax |
| 2 | train/No Pneumothorax/000002.dcm | No Pneumothorax |
| 3 | train/Pneumothorax/000003.dcm | Pneumothorax |
| 4 | train/Pneumothorax/000004.dcm | Pneumothorax |
现在,我们使用 DataBlock 类来准备 DICOM 数据以进行训练。
由于我们处理的是DICOM图像,因此需要将PILDicom用作ImageBlock类别。这样,DataBlock将知道如何打开DICOM图像。由于这是一个二分类任务,我们将使用CategoryBlock。
pneumothorax = DataBlock(blocks=(ImageBlock(cls=PILDicom), CategoryBlock),
get_x=lambda x:pneumothorax_source/f"{x[0]}",
get_y=lambda x:x[1],
batch_tfms=[*aug_transforms(size=224),Normalize.from_stats(*imagenet_stats)])
dls = pneumothorax.dataloaders(df.values, num_workers=0)此外,我们绘制了使用指定变换的第一批数据:
dls = pneumothorax.dataloaders(df.values)
dls.show_batch(max_n=16)Due to IPython and Windows limitation, python multiprocessing isn't available now.
So `number_workers` is changed to 0 to avoid getting stuck训练
我们可以使用 vision_learner 函数并开始训练。
learn = vision_learner(dls, resnet34, metrics=accuracy)注意,如果您不选择损失函数或优化器,fastai会尝试为该任务选择最佳的选项。您可以通过调用 loss_func 来检查损失函数。
learn.loss_funcFlattenedLoss of CrossEntropyLoss()您可以通过调用 opt_func 对优化器执行相同操作。
learn.opt_func<function fastai.optimizer.Adam(params, lr, mom=0.9, sqr_mom=0.99, eps=1e-05, wd=0.01, decouple_wd=True)>使用 lr_find 来尝试找到最佳的学习率
learn.lr_find()SuggestedLRs(lr_min=0.005754399299621582, lr_steep=0.0063095735386013985)learn.fit_one_cycle(1)| epoch | train_loss | valid_loss | accuracy | time |
|---|---|---|---|---|
| 0 | 1.191782 | 2.123666 | 0.320000 | 00:37 |
learn.predict(pneumothorax_source/f"train/Pneumothorax/000004.dcm")在对图像进行预测时,learn.predict 返回一个元组 (类别, 类别张量和 [每个类别的概率])。在这个数据集中只有 2 个类别 无肺气肿 和 肺气肿,因此每个概率有 2 个值,第一个值是图像属于 类别 0 或 无肺气肿 的概率,第二个值是图像属于 类别 1 或 肺气肿 的概率。
tta = learn.tta(use_max=True)learn.show_results(max_n=16)interp = Interpretation.from_learner(learn)interp.plot_top_losses(2)结果评估
医疗模型通常具有较高的影响力,因此了解一个模型在检测某种疾病时的表现至关重要。
该模型的准确率为56%。准确率可以定义为所有数据点中正确预测的数据点数量。然而,在此背景下,我们可以将准确率定义为模型正确且患者具有该病症的概率 加上 模型正确且患者没有该病症的概率。
在评估医疗模型时,还有一些其他关键术语需要使用:
假阳性与假阴性
假阳性是指一种错误,测试结果错误地表明存在某种状况,例如某种疾病(结果为阳性),而实际上并不存在。
假阴性是指一种错误,测试结果错误地表明不存在某种状况(结果为阴性),而实际上是存在的。
敏感性与特异性
- 敏感性或真正阳性率 是指模型在患者实际上确实患有该疾病的情况下将患者分类为有疾病的能力。敏感性量化了对假阴性的避免。
示例:一个新测试在10,000名患者中进行了测试,如果该新测试的敏感性为90%,则该测试将正确检测出9,000名(真正阳性)患者,但将漏掉1,000名(假阴性)实际患有该疾病但被测试为没有该疾病的患者。
- 特异性或真阴性率 是指模型在患者实际上并未患有该疾病的情况下将患者分类为没有疾病的能力。特异性量化了对假阳性的避免。
理解和使用敏感性、特异性和预测值 是一篇很好的论文,如果你有兴趣了解更多关于敏感性、特异性和预测值的知识。
阳性预测值和阴性预测值
大多数医学测试通过 PPV(阳性预测值)或 NPV(阴性预测值)进行评估。
PPV - 如果模型预测一个患者有某种疾病,那么该患者实际上有这种疾病的概率是多少。
NPV - 如果模型预测一个患者没有某种疾病,那么该患者实际上没有这种疾病的概率是多少。
PPV 的理想值是 1(100%),而最差的可能值是零。
NPV 的理想值是 1(100%),而最差的可能值是零。
混淆矩阵
混淆矩阵是针对valid数据集绘制的
interp = ClassificationInterpretation.from_learner(learn)
losses,idxs = interp.top_losses()
len(dls.valid_ds)==len(losses)==len(idxs)
interp.plot_confusion_matrix(figsize=(7,7))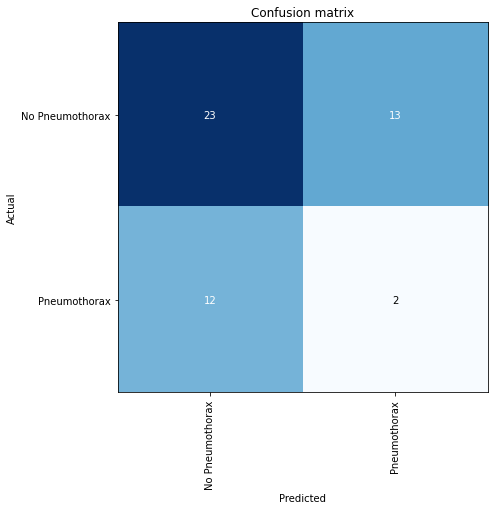
您也可以通过以下方式重现从plot_confusion_matrix解读的结果:
upp, low = interp.confusion_matrix()
tn, fp = upp[0], upp[1]
fn, tp = low[0], low[1]
print(tn, fp, fn, tp)23 13 12 2注意到 灵敏度 = 真阳性 / (真阳性 + 假阴性)
sensitivity = tp/(tp + fn)
sensitivity0.14285714285714285在这种情况下,模型的敏感性为40%,因此只能正确检测到40%的真正阳性(即确诊为气胸的患者),但会漏掉60%的假阴性(实际上患有气胸但被告知没有的患者!这可不是一个好的情况)。
这也被称为第二类错误
特异性 = 真阴性 / (假阳性 + 真阴性)
specificity = tn/(fp + tn)
specificity0.6388888888888888该模型的特异性为63%,因此能够正确检测到63%的时间内患者没有气胸,但会错误地将37%的患者分类为具有气胸(假阳性),实际上并没有。
这也称为I型错误
阳性预测值 (PPV)
ppv = tp/(tp+fp)
ppv0.13333333333333333在这种情况下,模型在正确预测患有气胸的患者方面表现不佳。
阴性预测值 (NPV)
npv = tn/(tn+fn)
npv0.6571428571428571该模型在预测没有气胸的患者方面更优秀。
计算准确率
该模型的准确性如前所述为56%,但这是如何计算的呢?我们可以将准确性视为:
准确率 = 敏感性 x 流行率 + 特异性 * (1 - 流行率)
其中患病率是一个统计概念,指的是在特定时间内,某个特定人群中存在的疾病病例数量。在这种情况下,患病率是指在有效数据集中,患有该疾病的患者数量与总患者数量的比例。
要查看有效数据集中的文件,可以调用 dls.valid_ds.cat。
val = dls.valid_ds.cat
#val[0]验证集中的肺气胸图像有15张(总共50张图像,可以通过 len(dls.valid_ds)检查),因此这里的发病率为15/50 = 0.3
prevalence = 15/50
prevalence0.3accuracy = (sensitivity * prevalence) + (specificity * (1 - prevalence))
accuracy0.490079365079365引用:
[1] Filice R 等人。利用机器学习对NIH胸部X光数据集进行气胸注释的众包。数字成像杂志 (2019)。 https://doi.org/10.1007/s10278-019-00299-9