Note
Go to the end to download the full example code. or to run this example in your browser via Binder
HDBSCAN聚类算法演示#
在本演示中,我们将从推广 cluster.DBSCAN 算法的角度来看 cluster.HDBSCAN 。
我们将在特定数据集上比较这两种算法。最后,我们将评估HDBSCAN对某些超参数的敏感性。
我们首先定义几个实用函数以方便使用。
import matplotlib.pyplot as plt
import numpy as np
from sklearn.cluster import DBSCAN, HDBSCAN
from sklearn.datasets import make_blobs
def plot(X, labels, probabilities=None, parameters=None, ground_truth=False, ax=None):
if ax is None:
_, ax = plt.subplots(figsize=(10, 4))
labels = labels if labels is not None else np.ones(X.shape[0])
probabilities = probabilities if probabilities is not None else np.ones(X.shape[0])
# 黑色被移除并用于噪声代替。
unique_labels = set(labels)
colors = [plt.cm.Spectral(each) for each in np.linspace(0, 1, len(unique_labels))]
# 一个点属于其标记簇的概率决定了其标记的大小
proba_map = {idx: probabilities[idx] for idx in range(len(labels))}
for k, col in zip(unique_labels, colors):
if k == -1:
# 黑色用于噪声。
col = [0, 0, 0, 1]
class_index = np.where(labels == k)[0]
for ci in class_index:
ax.plot(
X[ci, 0],
X[ci, 1],
"x" if k == -1 else "o",
markerfacecolor=tuple(col),
markeredgecolor="k",
markersize=4 if k == -1 else 1 + 5 * proba_map[ci],
)
n_clusters_ = len(set(labels)) - (1 if -1 in labels else 0)
preamble = "True" if ground_truth else "Estimated"
title = f"{preamble} number of clusters: {n_clusters_}"
if parameters is not None:
parameters_str = ", ".join(f"{k}={v}" for k, v in parameters.items())
title += f" | {parameters_str}"
ax.set_title(title)
plt.tight_layout()
生成样本数据#
HDBSCAN 相较于 DBSCAN 的最大优势之一是其开箱即用的鲁棒性。特别是在异质数据混合时表现尤为出色。与 DBSCAN 一样,它可以对任意形状和分布进行建模,但与 DBSCAN 不同的是,它不需要指定一个任意且敏感的 eps 超参数。
例如,下面我们从三个二维各向同性高斯分布的混合中生成一个数据集。
centers = [[1, 1], [-1, -1], [1.5, -1.5]]
X, labels_true = make_blobs(
n_samples=750, centers=centers, cluster_std=[0.4, 0.1, 0.75], random_state=0
)
plot(X, labels=labels_true, ground_truth=True)
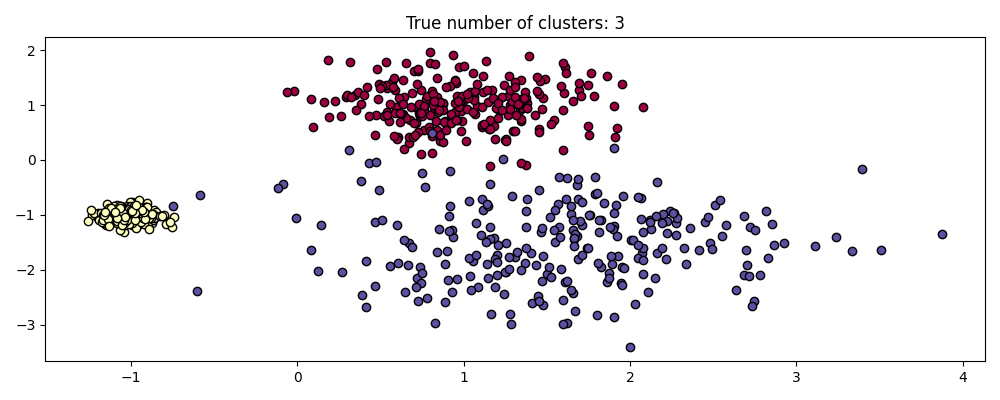
尺度不变性#
值得记住的是,虽然DBSCAN为 eps 参数提供了一个默认值,但它几乎没有一个合适的默认值,必须针对使用的特定数据集进行调整。
作为一个简单的示例,考虑对一个数据集调整 eps 值进行聚类,并将相同的值应用于数据集的重新缩放版本所获得的聚类。
fig, axes = plt.subplots(3, 1, figsize=(10, 12))
dbs = DBSCAN(eps=0.3)
for idx, scale in enumerate([1, 0.5, 3]):
dbs.fit(X * scale)
plot(X * scale, dbs.labels_, parameters={"scale": scale, "eps": 0.3}, ax=axes[idx])
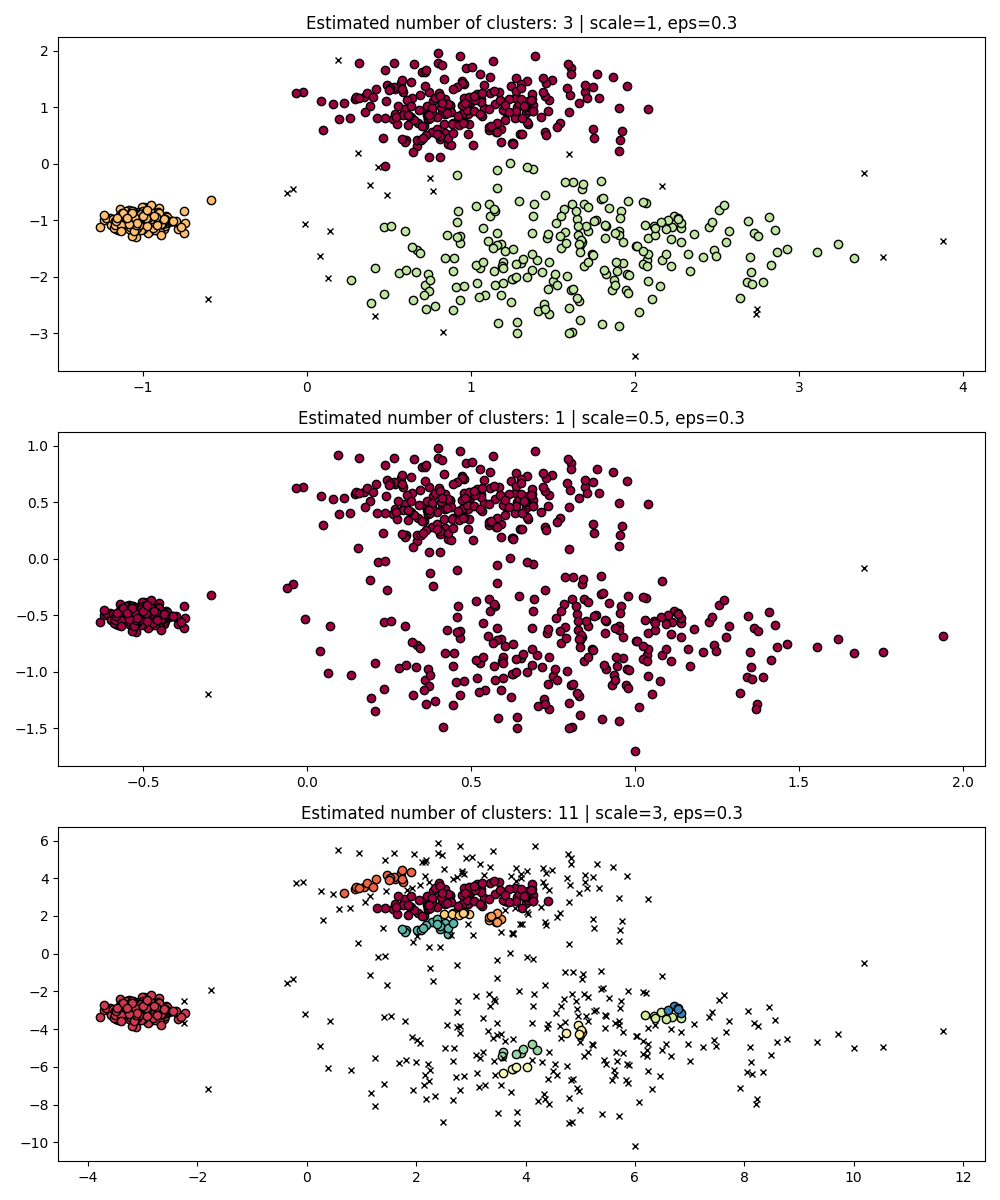
确实,为了保持相同的结果,我们必须按相同的比例缩放 eps 。
fig, axis = plt.subplots(1, 1, figsize=(12, 5))
dbs = DBSCAN(eps=0.9).fit(3 * X)
plot(3 * X, dbs.labels_, parameters={"scale": 3, "eps": 0.9}, ax=axis)
在标准化数据时(例如使用:class:sklearn.preprocessing.StandardScaler ),虽然有助于缓解这个问题,但必须非常小心地选择合适的 eps 值。
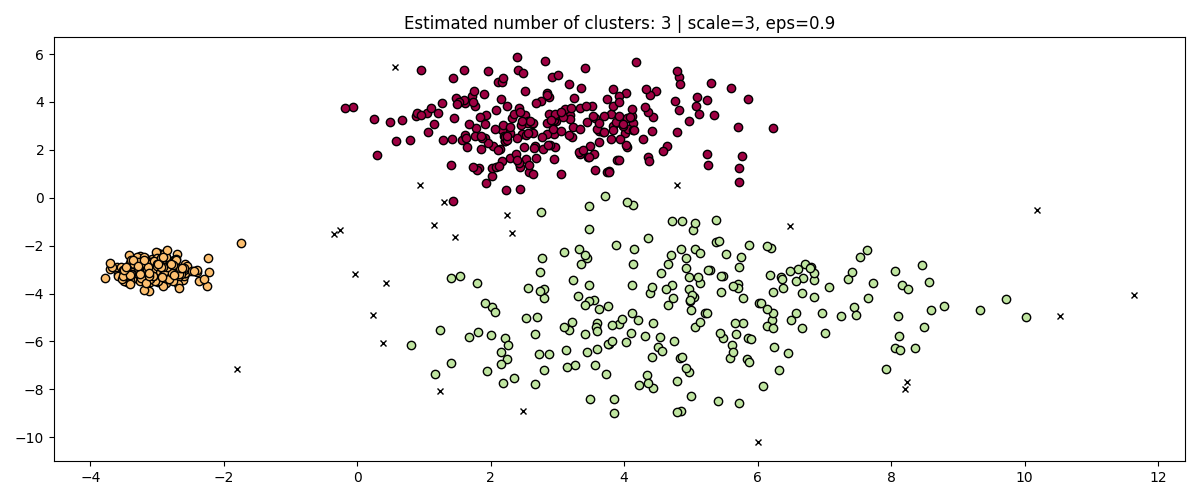
HDBSCAN 在这方面更加稳健:HDBSCAN 可以被视为对所有可能的 eps 值进行聚类,并从所有可能的聚类中提取最佳聚类(参见 用户指南 )。一个直接的优势是 HDBSCAN 是尺度不变的。
fig, axes = plt.subplots(3, 1, figsize=(10, 12))
hdb = HDBSCAN()
for idx, scale in enumerate([1, 0.5, 3]):
hdb.fit(X * scale)
plot(
X * scale,
hdb.labels_,
hdb.probabilities_,
ax=axes[idx],
parameters={"scale": scale},
)
多尺度聚类#
HDBSCAN 不仅仅是尺度不变的,它还能够进行多尺度聚类,能够处理密度不同的簇。传统的 DBSCAN 假设任何潜在的簇在密度上是均匀的。HDBSCAN 不受这种限制。为了演示这一点,我们考虑以下数据集
centers = [[-0.85, -0.85], [-0.85, 0.85], [3, 3], [3, -3]]
X, labels_true = make_blobs(
n_samples=750, centers=centers, cluster_std=[0.2, 0.35, 1.35, 1.35], random_state=0
)
plot(X, labels=labels_true, ground_truth=True)
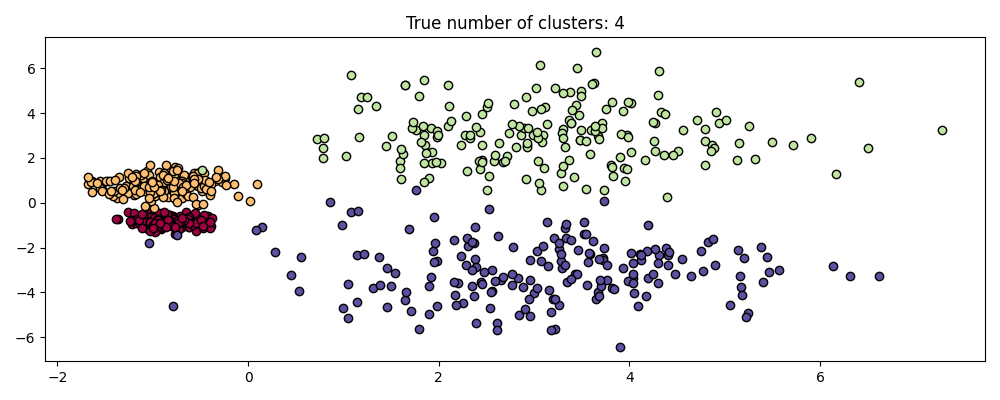
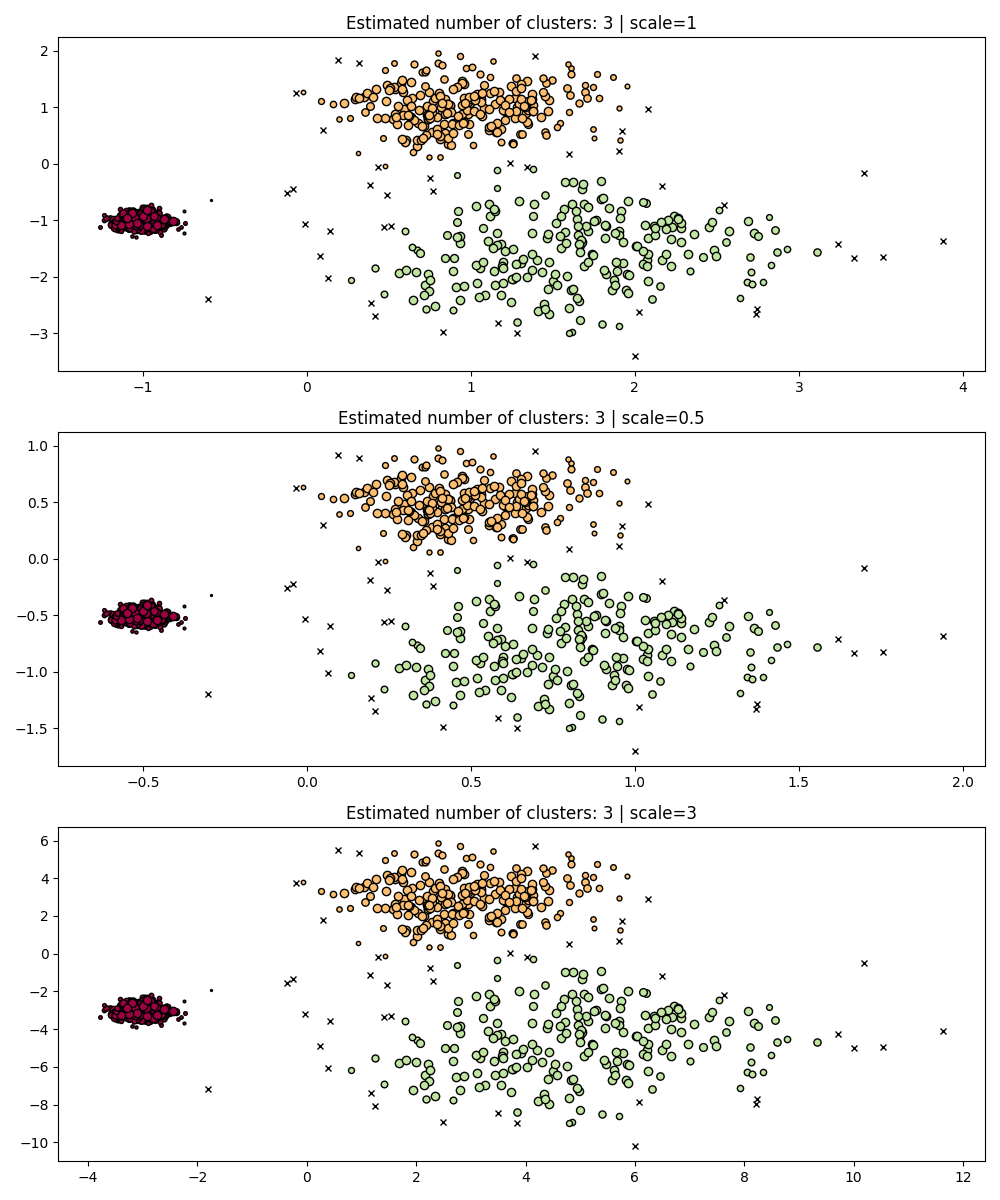
这个数据集对于DBSCAN来说更具挑战性,因为它具有不同的密度和空间分离:
如果
eps过大,我们可能会错误地将两个密集簇聚为一个,因为它们的相互可达性会扩展簇。如果
eps过小,我们可能会将稀疏簇分裂成许多错误的簇。
更不用说,这还需要手动调整 eps 的选择,直到我们找到一个我们满意的折衷方案。
fig, axes = plt.subplots(2, 1, figsize=(10, 8))
params = {"eps": 0.7}
dbs = DBSCAN(**params).fit(X)
plot(X, dbs.labels_, parameters=params, ax=axes[0])
params = {"eps": 0.3}
dbs = DBSCAN(**params).fit(X)
plot(X, dbs.labels_, parameters=params, ax=axes[1])
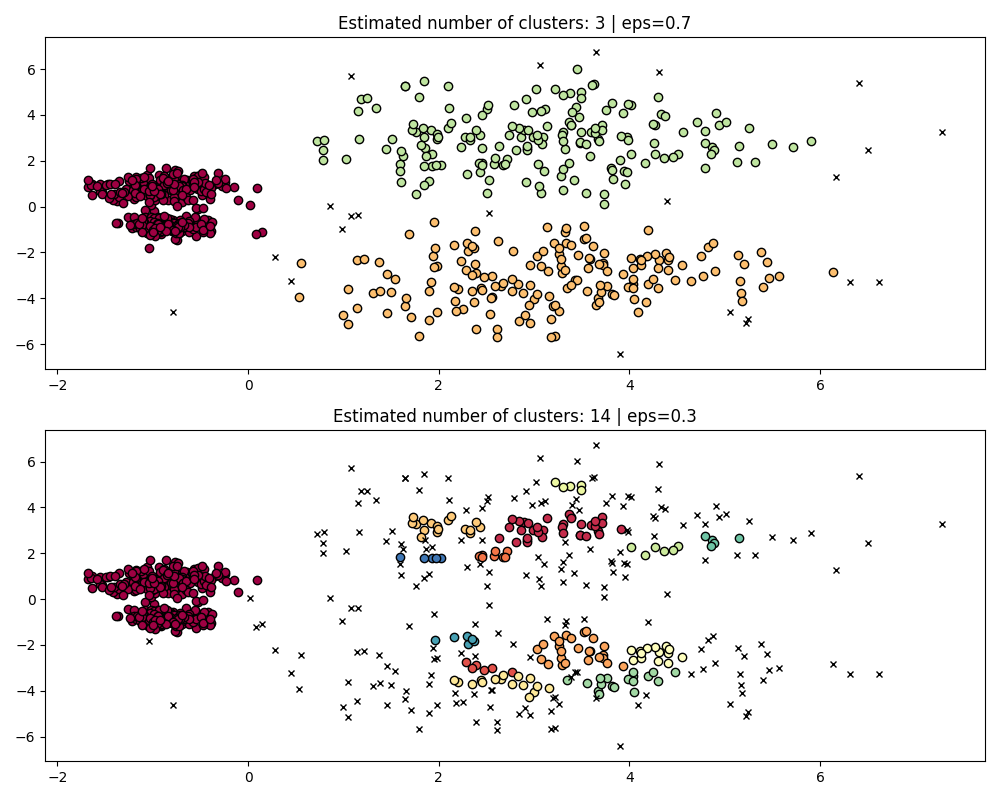
为了正确聚类这两个密集的簇,我们需要一个更小的epsilon值,然而在 eps=0.3 时,我们已经在分裂稀疏簇,随着epsilon的减小,这种情况只会变得更加严重。实际上,DBSCAN似乎无法在同时分离两个密集簇的同时防止稀疏簇的分裂。让我们与HDBSCAN进行比较。
hdb = HDBSCAN().fit(X)
plot(X, hdb.labels_, hdb.probabilities_)
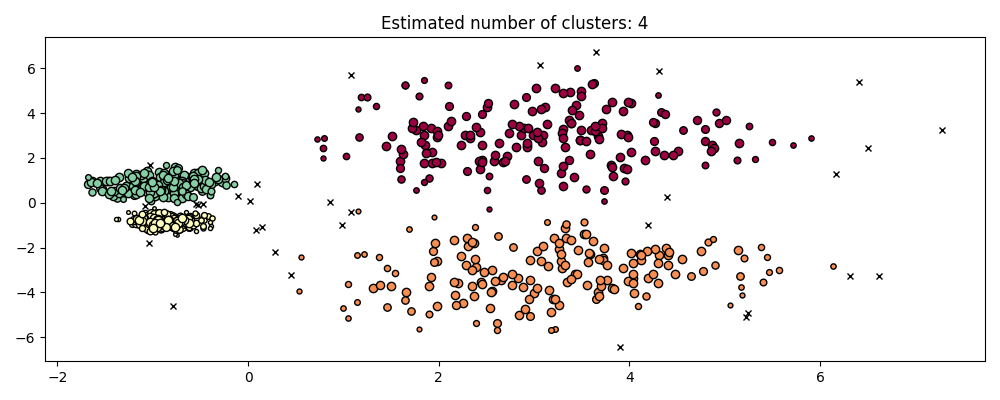
HDBSCAN能够适应数据集的多尺度结构而无需参数调整。虽然任何足够有趣的数据集都需要调整参数,但这种情况下,HDBSCAN可以在无需用户干预的情况下生成质量更好的聚类结果,而这些结果是DBSCAN无法实现的。
超参数鲁棒性#
在任何实际应用中,超参数调优最终都是一个重要步骤,因此让我们来看看 HDBSCAN 的一些最重要的超参数。虽然 HDBSCAN 不需要 DBSCAN 的 eps 参数,但它仍然有一些超参数,如 min_cluster_size 和 min_samples ,这些参数可以调整其关于密度的结果。然而,我们将看到,得益于这些参数的明确含义,HDBSCAN 对各种实际示例具有相对的鲁棒性。
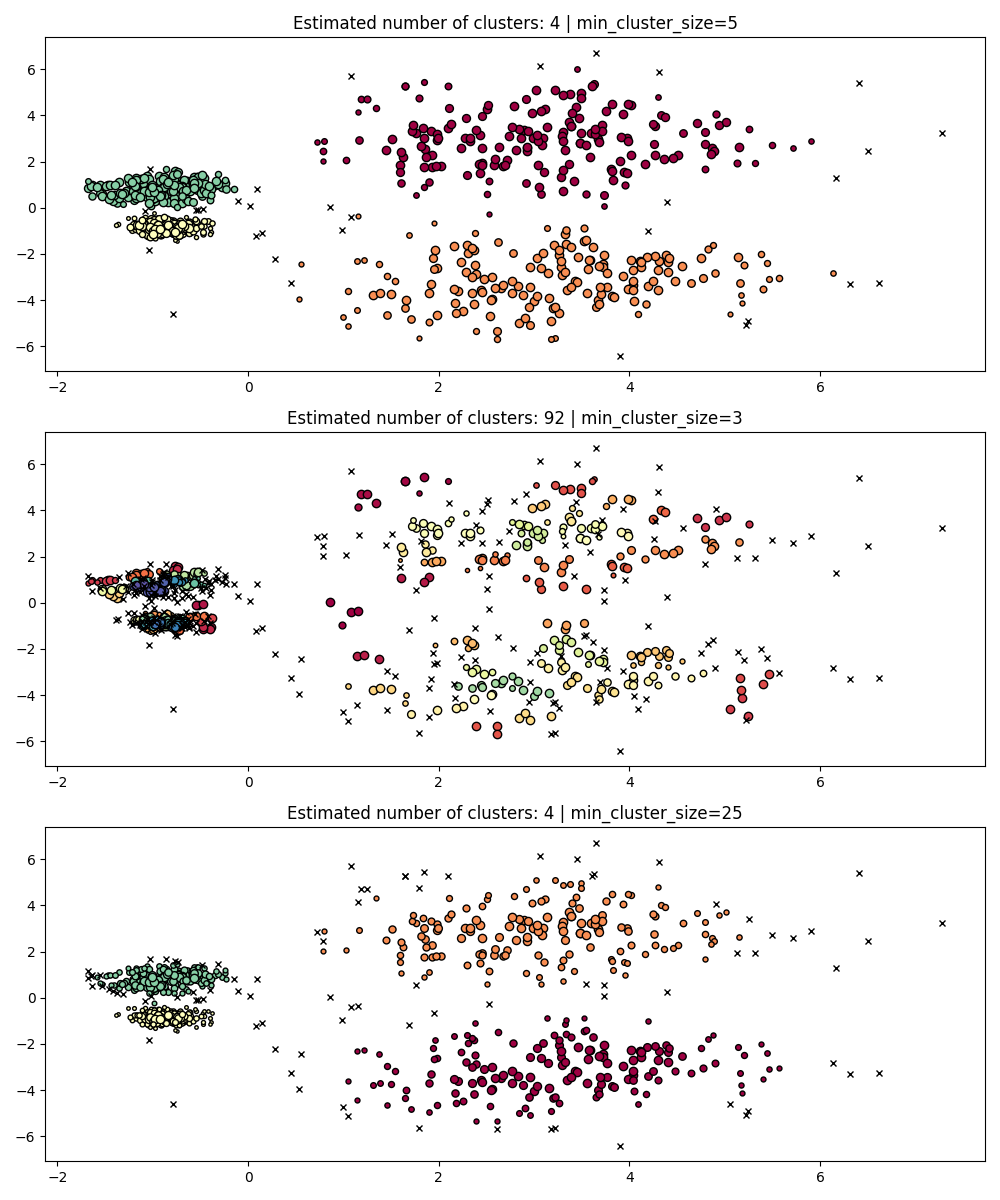
min_cluster_size 是一个组被认为是簇所需的最小样本数量。
小于此大小的簇将被视为噪声。默认值为5。此参数通常根据需要调整为更大的值。较小的值可能会导致较少的点被标记为噪声。然而,过小的值会导致错误的子簇被识别和偏好。较大的值在处理噪声数据集时往往更为稳健,例如具有显著重叠的高方差簇。
PARAM = ({"min_cluster_size": 5}, {"min_cluster_size": 3}, {"min_cluster_size": 25})
fig, axes = plt.subplots(3, 1, figsize=(10, 12))
for i, param in enumerate(PARAM):
hdb = HDBSCAN(**param).fit(X)
labels = hdb.labels_
plot(X, labels, hdb.probabilities_, param, ax=axes[i])
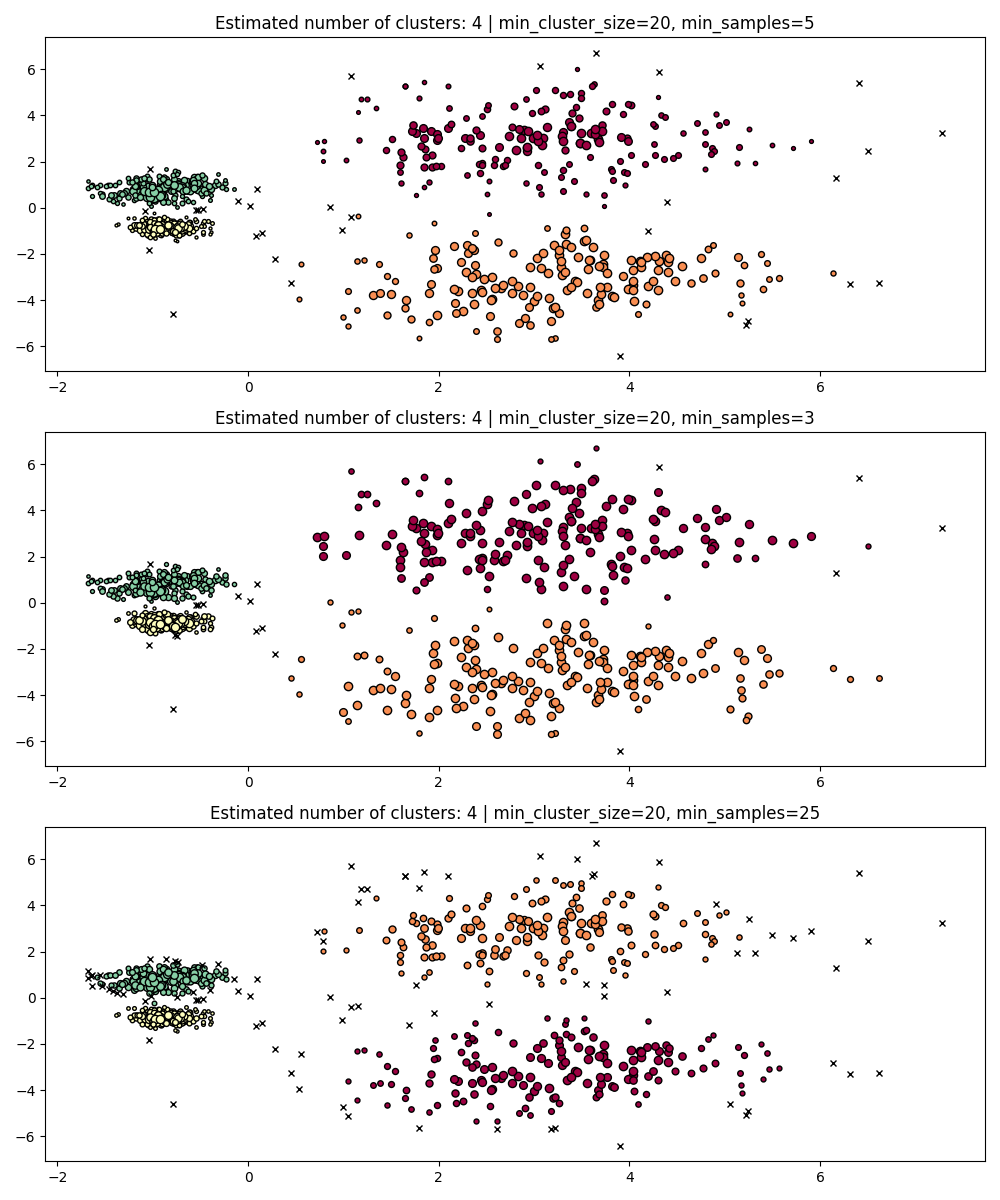
min_samples#
min_samples 是一个点被认为是核心点的邻域内样本数量,包括该点本身。
min_samples 默认为 min_cluster_size 。
与 min_cluster_size 类似,较大的 min_samples 值会增加模型对噪声的鲁棒性,但有可能忽略或丢弃潜在的有效但较小的簇。
在找到 min_cluster_size 的合适值后,最好再调整 min_samples 。
PARAM = (
{"min_cluster_size": 20, "min_samples": 5},
{"min_cluster_size": 20, "min_samples": 3},
{"min_cluster_size": 20, "min_samples": 25},
)
fig, axes = plt.subplots(3, 1, figsize=(10, 12))
for i, param in enumerate(PARAM):
hdb = HDBSCAN(**param).fit(X)
labels = hdb.labels_
plot(X, labels, hdb.probabilities_, param, ax=axes[i])
dbscan_clustering#
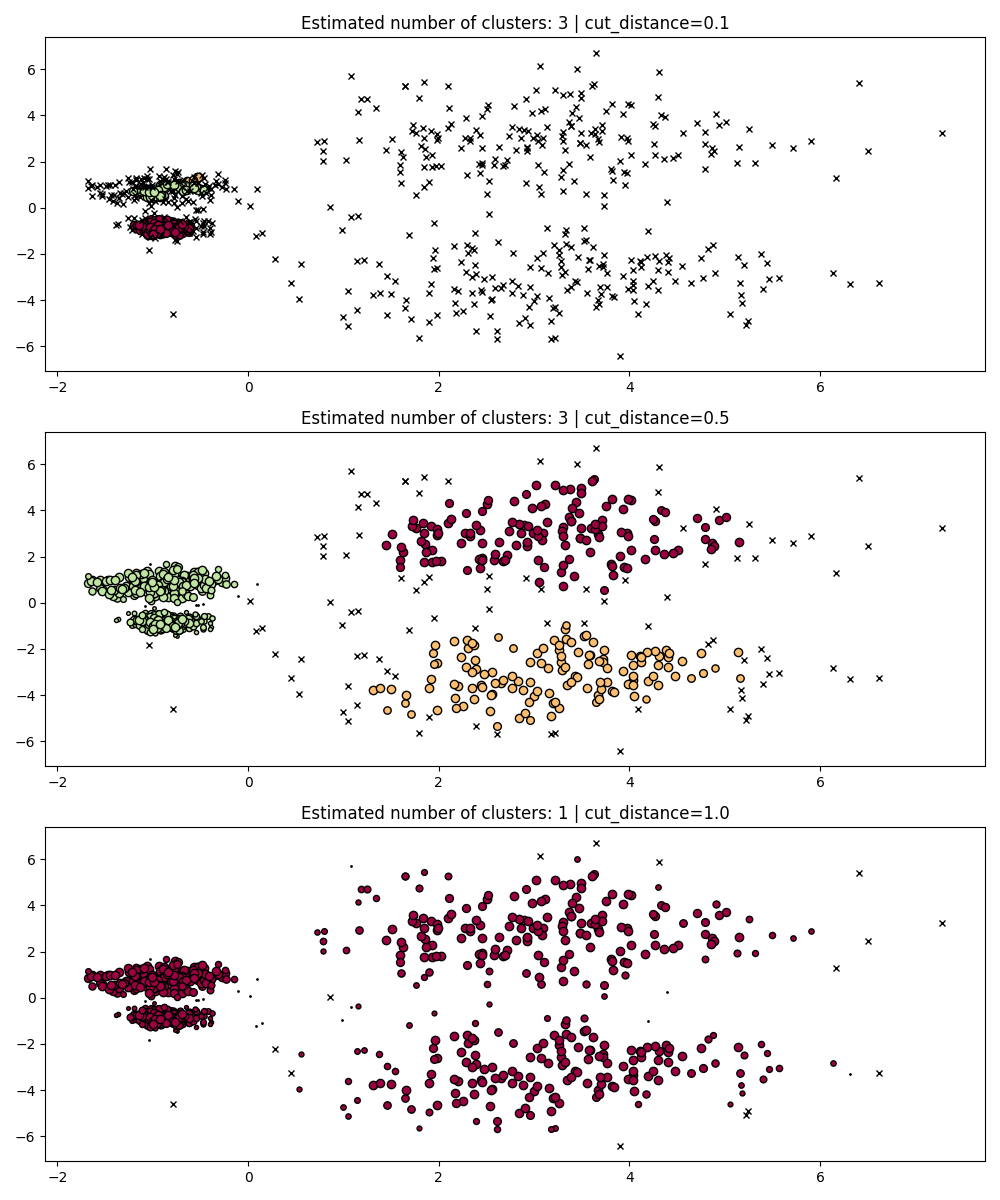
在 fit 过程中, HDBSCAN 构建了一棵单链树,该树编码了所有点在 DBSCAN 的 eps 参数下的聚类。
因此,我们可以高效地绘制和评估这些聚类,而无需完全重新计算核心距离、互达性和最小生成树等中间值。我们只需要指定我们想要聚类的 cut_distance (相当于 eps )即可。
PARAM = (
{"cut_distance": 0.1},
{"cut_distance": 0.5},
{"cut_distance": 1.0},
)
hdb = HDBSCAN()
hdb.fit(X)
fig, axes = plt.subplots(len(PARAM), 1, figsize=(10, 12))
for i, param in enumerate(PARAM):
labels = hdb.dbscan_clustering(**param)
plot(X, labels, hdb.probabilities_, param, ax=axes[i])
Total running time of the script: (0 minutes 5.864 seconds)
Related examples