Note
Go to the end to download the full example code. or to run this example in your browser via Binder
使用邻域成分分析进行降维#
使用邻域成分分析进行降维的示例。
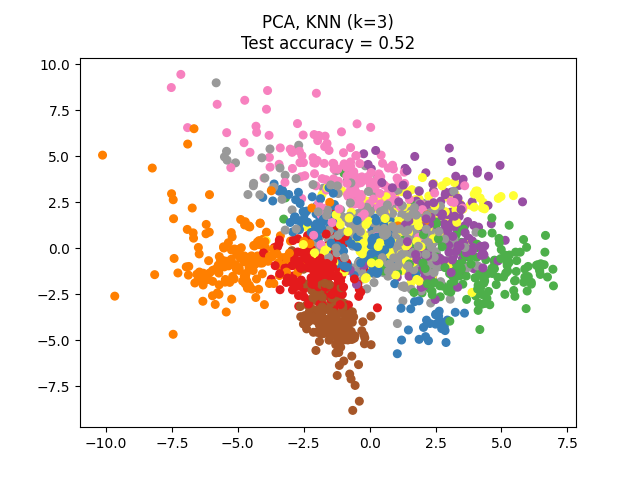
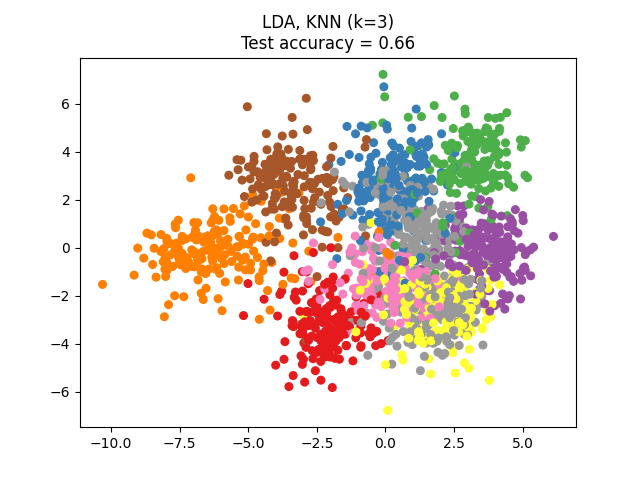
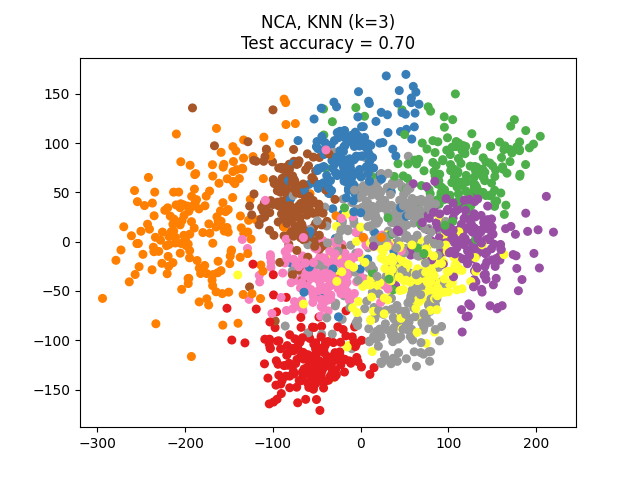
本示例比较了不同的(线性)降维方法在数字数据集上的应用。该数据集包含从0到9的数字图像,每个类别大约有180个样本。每张图像的维度为8x8 = 64,并被降维到二维数据点。
主成分分析(PCA)应用于此数据集,识别出在数据中占最大方差的属性组合(主成分,或特征空间中的方向)。在这里,我们将不同的样本绘制在前两个主成分上。
线性判别分析(LDA)试图识别在*类别之间*占最大方差的属性。特别地,LDA与PCA不同,是一种有监督的方法,使用已知的类别标签。
邻域成分分析(NCA)试图找到一个特征空间,使得随机最近邻算法能够提供最佳的准确性。与LDA一样,它也是一种有监督的方法。
可以看到,尽管维度大幅度降低,NCA仍然强制数据进行视觉上有意义的聚类。
# SPDX-License-Identifier: BSD-3-Clause
import matplotlib.pyplot as plt
import numpy as np
from sklearn import datasets
from sklearn.decomposition import PCA
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis
from sklearn.model_selection import train_test_split
from sklearn.neighbors import KNeighborsClassifier, NeighborhoodComponentsAnalysis
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import StandardScaler
n_neighbors = 3
random_state = 0
# 加载数字数据集
X, y = datasets.load_digits(return_X_y=True)
# 划分为训练集/测试集
X_train, X_test, y_train, y_test = train_test_split(
X, y, test_size=0.5, stratify=y, random_state=random_state
)
dim = len(X[0])
n_classes = len(np.unique(y))
# 使用PCA将维度减少到2
pca = make_pipeline(StandardScaler(), PCA(n_components=2, random_state=random_state))
# 使用线性判别分析将维度减少到2
lda = make_pipeline(StandardScaler(), LinearDiscriminantAnalysis(n_components=2))
# 使用邻域成分分析将维度减少到2
nca = make_pipeline(
StandardScaler(),
NeighborhoodComponentsAnalysis(n_components=2, random_state=random_state),
)
# 使用最近邻分类器来评估这些方法
knn = KNeighborsClassifier(n_neighbors=n_neighbors)
# 列出要比较的方法
dim_reduction_methods = [("PCA", pca), ("LDA", lda), ("NCA", nca)]
# plt.figure()
for i, (name, model) in enumerate(dim_reduction_methods):
plt.figure()
# plt.subplot(1, 3, i + 1, aspect=1)
# 拟合该方法的模型
model.fit(X_train, y_train)
# 在嵌入的训练集上拟合一个最近邻分类器
knn.fit(model.transform(X_train), y_train)
# 计算嵌入测试集上的最近邻准确率
acc_knn = knn.score(model.transform(X_test), y_test)
# 使用拟合模型将数据集嵌入到二维空间中
X_embedded = model.transform(X)
# 绘制投影点并显示评估分数
plt.scatter(X_embedded[:, 0], X_embedded[:, 1], c=y, s=30, cmap="Set1")
plt.title(
"{}, KNN (k={})\nTest accuracy = {:.2f}".format(name, n_neighbors, acc_knn)
)
plt.show()
Total running time of the script: (0 minutes 0.967 seconds)
Related examples