Note
Go to the end to download the full example code. or to run this example in your browser via Binder
异常检测估计器的评估#
本示例比较了两种异常检测算法,即局部异常因子(LOF)和孤立森林(IForest),在
sklearn.datasets 中提供的真实世界数据集上的表现。目的是展示不同算法在不同数据集上的表现,并对比它们的训练速度和对超参数的敏感性。
这些算法在假定包含异常值的整个数据集上进行训练(不使用标签)。
使用已知的真实标签计算并显示ROC曲线,使用
RocCurveDisplay。通过ROC-AUC评估性能。
# 作者:scikit-learn 开发者
# SPDX 许可证标识符:BSD-3-Clause
数据集预处理和模型训练#
不同的异常值检测模型需要不同的预处理。在存在分类变量的情况下,OrdinalEncoder 通常是基于树的模型(如 IsolationForest )的一个好策略,而基于邻居的模型(如 LocalOutlierFactor )则会受到序数编码引入的顺序的影响。为了避免引入顺序,应该使用 OneHotEncoder 。
基于邻居的模型可能还需要对数值特征进行缩放(参见 k-邻近模型的重新缩放效果 )。在存在异常值的情况下,一个好的选择是使用 RobustScaler 。
from sklearn.compose import ColumnTransformer
from sklearn.ensemble import IsolationForest
from sklearn.neighbors import LocalOutlierFactor
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import (
OneHotEncoder,
OrdinalEncoder,
RobustScaler,
)
def make_estimator(name, categorical_columns=None, iforest_kw=None, lof_kw=None):
"""基于其名称创建一个异常值检测估计器。"""
if name == "LOF":
outlier_detector = LocalOutlierFactor(**(lof_kw or {}))
if categorical_columns is None:
preprocessor = RobustScaler()
else:
preprocessor = ColumnTransformer(
transformers=[("categorical", OneHotEncoder(), categorical_columns)],
remainder=RobustScaler(),
)
else: # name == "IForest"
outlier_detector = IsolationForest(**(iforest_kw or {}))
if categorical_columns is None:
preprocessor = None
else:
ordinal_encoder = OrdinalEncoder(
handle_unknown="use_encoded_value", unknown_value=-1
)
preprocessor = ColumnTransformer(
transformers=[
("categorical", ordinal_encoder, categorical_columns),
],
remainder="passthrough",
)
return make_pipeline(preprocessor, outlier_detector)
以下 fit_predict 函数返回 X 的平均异常值得分。
from time import perf_counter
def fit_predict(estimator, X):
tic = perf_counter()
if estimator[-1].__class__.__name__ == "LocalOutlierFactor":
estimator.fit(X)
y_pred = estimator[-1].negative_outlier_factor_
else: # "IsolationForest"
y_pred = estimator.fit(X).decision_function(X)
toc = perf_counter()
print(f"Duration for {model_name}: {toc - tic:.2f} s")
return y_pred
在接下来的示例中,我们每个部分处理一个数据集。加载数据后,目标被修改为包含两个类别:0 代表正常值,1 代表异常值。由于 scikit-learn 文档的计算限制,某些数据集的样本量使用分层的 train_test_split 进行了缩减。
此外,我们将 n_neighbors 设置为与预期的异常数量 expected_n_anomalies = n_samples * expected_anomaly_fraction 相匹配。这是一个很好的启发式方法,只要异常值的比例不是非常低,原因是 n_neighbors 至少应该大于样本数量较少的簇中的样本数量(参见 使用局部离群因子(LOF)进行离群点检测 )。
KDDCup99 - SA 数据集#
Kddcup 99 dataset 是在一个封闭网络中生成的,并且手动注入了攻击。SA 数据集是它的一个子集,通过简单地选择所有正常数据和大约 3% 的异常比例获得。
import numpy as np
from sklearn.datasets import fetch_kddcup99
from sklearn.model_selection import train_test_split
X, y = fetch_kddcup99(
subset="SA", percent10=True, random_state=42, return_X_y=True, as_frame=True
)
y = (y != b"normal.").astype(np.int32)
X, _, y, _ = train_test_split(X, y, train_size=0.1, stratify=y, random_state=42)
n_samples, anomaly_frac = X.shape[0], y.mean()
print(f"{n_samples} datapoints with {y.sum()} anomalies ({anomaly_frac:.02%})")
10065 datapoints with 338 anomalies (3.36%)
SA 数据集包含 41 个特征,其中 3 个是分类特征:”protocol_type”、”service” 和 “flag”。
y_true = {}
y_pred = {"LOF": {}, "IForest": {}}
model_names = ["LOF", "IForest"]
cat_columns = ["protocol_type", "service", "flag"]
y_true["KDDCup99 - SA"] = y
for model_name in model_names:
model = make_estimator(
name=model_name,
categorical_columns=cat_columns,
lof_kw={"n_neighbors": int(n_samples * anomaly_frac)},
iforest_kw={"random_state": 42},
)
y_pred[model_name]["KDDCup99 - SA"] = fit_predict(model, X)
Duration for LOF: 0.69 s
Duration for IForest: 0.20 s
森林覆盖类型数据集#
Forest covertypes 是一个多分类数据集,其中目标是给定森林区域的主要树种。它包含54个特征,其中一些特征(“Wilderness_Area”和“Soil_Type”)已经进行了二进制编码。虽然最初是作为分类任务设计的,但可以将标签为2的样本视为内点,将标签为4的样本视为外点。
from sklearn.datasets import fetch_covtype
X, y = fetch_covtype(return_X_y=True, as_frame=True)
s = (y == 2) + (y == 4)
X = X.loc[s]
y = y.loc[s]
y = (y != 2).astype(np.int32)
X, _, y, _ = train_test_split(X, y, train_size=0.05, stratify=y, random_state=42)
X_forestcover = X # save X for later use
n_samples, anomaly_frac = X.shape[0], y.mean()
print(f"{n_samples} datapoints with {y.sum()} anomalies ({anomaly_frac:.02%})")
14302 datapoints with 137 anomalies (0.96%)
y_true["forestcover"] = y
for model_name in model_names:
model = make_estimator(
name=model_name,
lof_kw={"n_neighbors": int(n_samples * anomaly_frac)},
iforest_kw={"random_state": 42},
)
y_pred[model_name]["forestcover"] = fit_predict(model, X)
Duration for LOF: 0.47 s
Duration for IForest: 0.15 s
阿姆斯房价数据集#
Ames住房数据集 最初是一个回归数据集,其目标是爱荷华州艾姆斯市房屋的销售价格。在这里,我们将其转换为一个异常检测问题,认为每平方英尺价格超过70美元的房屋为异常。为了简化问题,我们去掉了每平方英尺价格在40到70美元之间的房屋。
import matplotlib.pyplot as plt
from sklearn.datasets import fetch_openml
X, y = fetch_openml(name="ames_housing", version=1, return_X_y=True, as_frame=True)
y = y.div(X["Lot_Area"])
# pandas 1.5.1 中的 None 值在 pandas 2.0.1 中被映射为 np.nan
X["Misc_Feature"] = X["Misc_Feature"].cat.add_categories("NoInfo").fillna("NoInfo")
X["Mas_Vnr_Type"] = X["Mas_Vnr_Type"].cat.add_categories("NoInfo").fillna("NoInfo")
X.drop(columns="Lot_Area", inplace=True)
mask = (y < 40) | (y > 70)
X = X.loc[mask]
y = y.loc[mask]
y.hist(bins=20, edgecolor="black")
plt.xlabel("House price in USD/sqft")
_ = plt.title("Distribution of house prices in Ames")
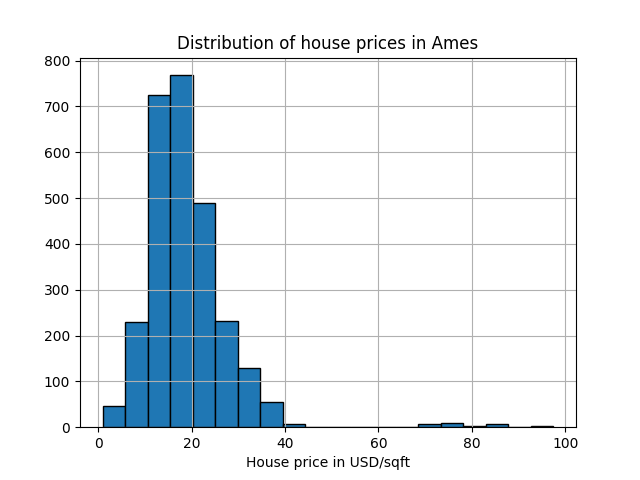
y = (y > 70).astype(np.int32)
n_samples, anomaly_frac = X.shape[0], y.mean()
print(f"{n_samples} datapoints with {y.sum()} anomalies ({anomaly_frac:.02%})")
2714 datapoints with 30 anomalies (1.11%)
该数据集包含46个分类特征。在这种情况下,使用:class:~sklearn.compose.make_column_selector 来查找它们比手动传递列表更容易。
from sklearn.compose import make_column_selector as selector
categorical_columns_selector = selector(dtype_include="category")
cat_columns = categorical_columns_selector(X)
y_true["ames_housing"] = y
for model_name in model_names:
model = make_estimator(
name=model_name,
categorical_columns=cat_columns,
lof_kw={"n_neighbors": int(n_samples * anomaly_frac)},
iforest_kw={"random_state": 42},
)
y_pred[model_name]["ames_housing"] = fit_predict(model, X)
Duration for LOF: 0.76 s
Duration for IForest: 0.14 s
心电图数据集#
Cardiotocography 数据集 是一个多类数据集,包含胎儿心电图(FHR)模式,类别用1到10的标签编码。这里我们将类别3(少数类)设为异常值。该数据集包含30个数值特征,其中一些是二进制编码的,一些是连续的。
X, y = fetch_openml(name="cardiotocography", version=1, return_X_y=True, as_frame=False)
X_cardiotocography = X # save X for later use
s = y == "3"
y = s.astype(np.int32)
n_samples, anomaly_frac = X.shape[0], y.mean()
print(f"{n_samples} datapoints with {y.sum()} anomalies ({anomaly_frac:.02%})")
2126 datapoints with 53 anomalies (2.49%)
y_true["cardiotocography"] = y
for model_name in model_names:
model = make_estimator(
name=model_name,
lof_kw={"n_neighbors": int(n_samples * anomaly_frac)},
iforest_kw={"random_state": 42},
)
y_pred[model_name]["cardiotocography"] = fit_predict(model, X)
Duration for LOF: 0.06 s
Duration for IForest: 0.08 s
绘制并解释结果#
算法性能与在低假阳性率(FPR)下的真阳性率(TPR)表现相关。最好的算法在图表的左上方,并且曲线下面积(AUC)接近1。对角虚线表示对异常值和正常值的随机分类。
import math
from sklearn.metrics import RocCurveDisplay
cols = 2
pos_label = 0 # mean 0 belongs to positive class
datasets_names = y_true.keys()
rows = math.ceil(len(datasets_names) / cols)
fig, axs = plt.subplots(nrows=rows, ncols=cols, squeeze=False, figsize=(10, rows * 4))
for ax, dataset_name in zip(axs.ravel(), datasets_names):
for model_idx, model_name in enumerate(model_names):
display = RocCurveDisplay.from_predictions(
y_true[dataset_name],
y_pred[model_name][dataset_name],
pos_label=pos_label,
name=model_name,
ax=ax,
plot_chance_level=(model_idx == len(model_names) - 1),
chance_level_kw={"linestyle": ":"},
)
ax.set_title(dataset_name)
_ = plt.tight_layout(pad=2.0) # spacing between subplots
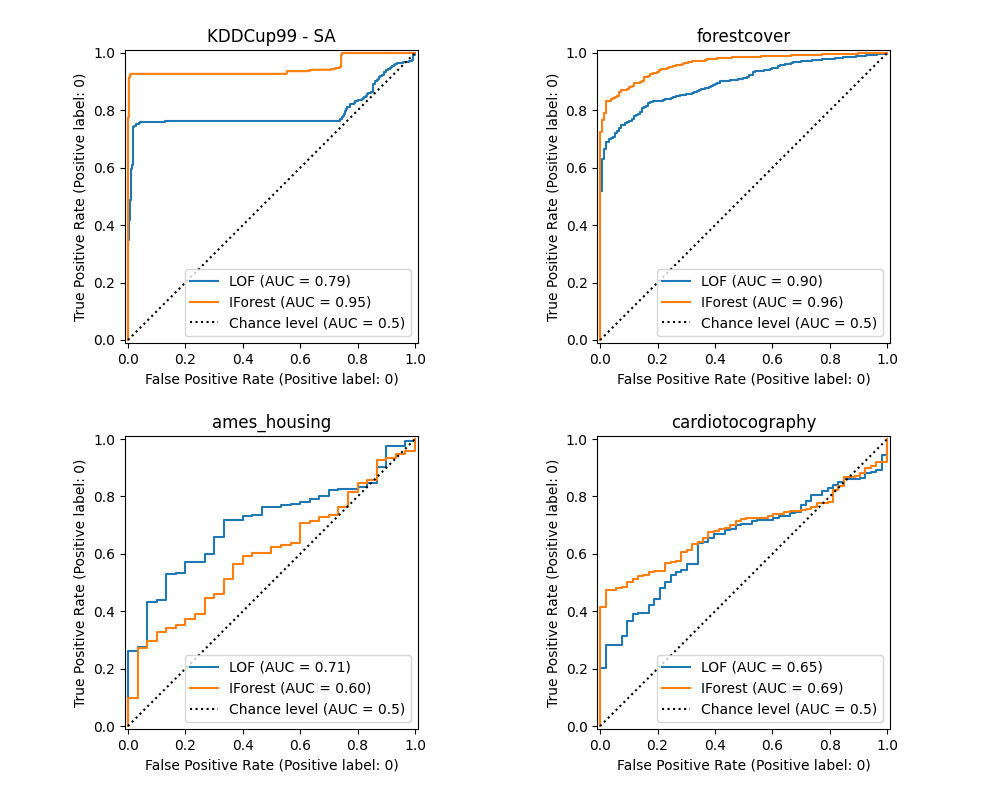
我们观察到,一旦调整了邻居数量,LOF 和 IForest 在 forestcover 和 cardiotocography 数据集的 ROC AUC 方面表现相似。对于 SA 数据集,IForest 的得分略好一些,而在 Ames housing 数据集上,LOF 的表现明显优于 IForest。
请注意,Isolation Forest 在处理包含大量样本的数据集时,训练速度通常比 LOF 快得多。LOF 需要计算成对距离以找到最近邻,这使得其复杂度与观测数量呈二次关系。这可能使得该方法在处理大型数据集时变得不可行。
消融研究#
在本节中,我们探讨超参数 n_neighbors 和数值变量缩放选择对 LOF 模型的影响。这里我们使用 Forest covertypes 数据集,因为二进制编码的类别在 0 和 1 之间引入了自然的欧几里得距离尺度。我们希望采用一种缩放方法,以避免给予非二进制特征特权,并且这种方法对异常值具有足够的鲁棒性,以使找到它们的任务不会变得过于困难。
X = X_forestcover
y = y_true["forestcover"]
n_samples = X.shape[0]
n_neighbors_list = (n_samples * np.array([0.2, 0.02, 0.01, 0.001])).astype(np.int32)
model = make_pipeline(RobustScaler(), LocalOutlierFactor())
linestyles = ["solid", "dashed", "dashdot", ":", (5, (10, 3))]
fig, ax = plt.subplots()
for model_idx, (linestyle, n_neighbors) in enumerate(zip(linestyles, n_neighbors_list)):
model.set_params(localoutlierfactor__n_neighbors=n_neighbors)
model.fit(X)
y_pred = model[-1].negative_outlier_factor_
display = RocCurveDisplay.from_predictions(
y,
y_pred,
pos_label=pos_label,
name=f"n_neighbors = {n_neighbors}",
ax=ax,
plot_chance_level=(model_idx == len(n_neighbors_list) - 1),
chance_level_kw={"linestyle": (0, (1, 10))},
linestyle=linestyle,
linewidth=2,
)
_ = ax.set_title("RobustScaler with varying n_neighbors\non forestcover dataset")
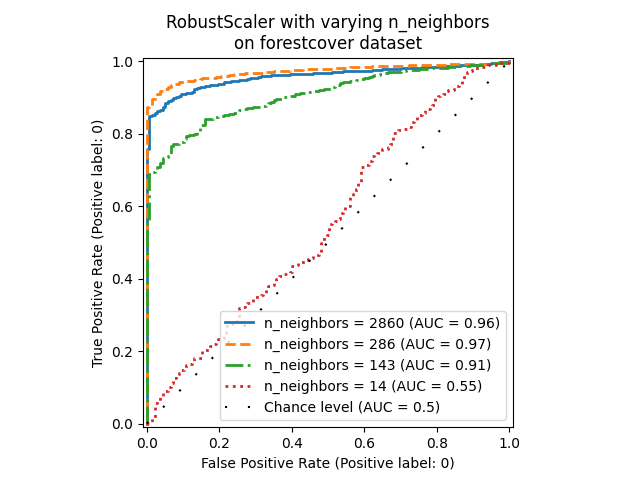
我们观察到邻居数量对模型性能有很大影响。如果可以获得(至少一些)真实标签,那么调整 n_neighbors 是很重要的。一种方便的方法是探索 n_neighbors 的值,其数量级与预期的污染程度相当。
from sklearn.preprocessing import MinMaxScaler, SplineTransformer, StandardScaler
preprocessor_list = [
None,
RobustScaler(),
StandardScaler(),
MinMaxScaler(),
SplineTransformer(),
]
expected_anomaly_fraction = 0.02
lof = LocalOutlierFactor(n_neighbors=int(n_samples * expected_anomaly_fraction))
fig, ax = plt.subplots()
for model_idx, (linestyle, preprocessor) in enumerate(
zip(linestyles, preprocessor_list)
):
model = make_pipeline(preprocessor, lof)
model.fit(X)
y_pred = model[-1].negative_outlier_factor_
display = RocCurveDisplay.from_predictions(
y,
y_pred,
pos_label=pos_label,
name=str(preprocessor).split("(")[0],
ax=ax,
plot_chance_level=(model_idx == len(preprocessor_list) - 1),
chance_level_kw={"linestyle": (0, (1, 10))},
linestyle=linestyle,
linewidth=2,
)
_ = ax.set_title("Fixed n_neighbors with varying preprocessing\non forestcover dataset")
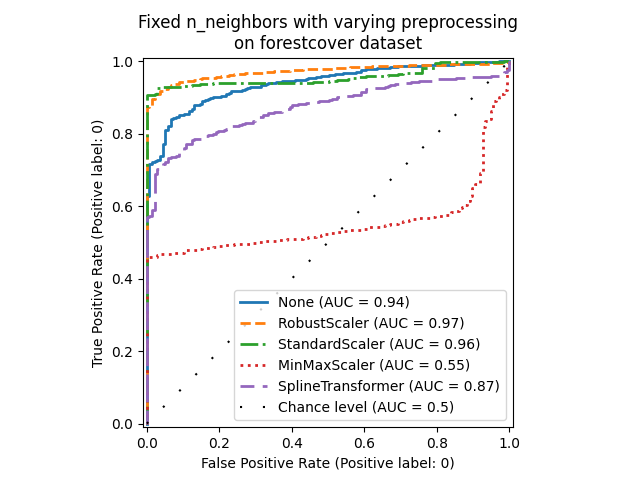
一方面,RobustScaler 默认使用四分位距(IQR)对每个特征进行独立缩放,四分位距是数据的第25百分位数和第75百分位数之间的范围。它通过减去中位数来中心化数据,然后通过除以IQR来缩放数据。IQR对离群值具有鲁棒性:与范围、均值和标准差相比,中位数和四分位距受极值的影响较小。此外,RobustScaler 不会像 StandardScaler 那样压缩边缘离群值。
另一方面,MinMaxScaler 会对每个特征单独进行缩放,使其范围映射到零和一之间。如果数据中存在异常值,它们可能会将数据偏向最小值或最大值,导致数据分布完全不同,并出现大的边缘异常值:所有非异常值可能会因此几乎聚集在一起。
我们还评估了完全不进行预处理(通过将 None 传递给管道),StandardScaler 和 SplineTransformer 。请参阅它们各自的文档以获取更多详细信息。
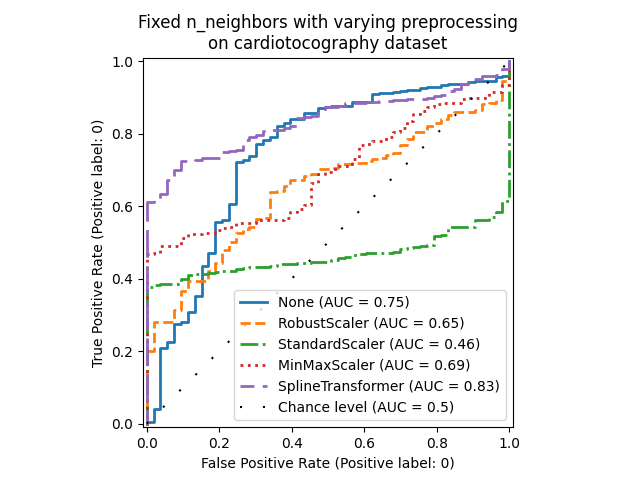
请注意,最佳预处理取决于数据集,如下所示:
X = X_cardiotocography
y = y_true["cardiotocography"]
n_samples, expected_anomaly_fraction = X.shape[0], 0.025
lof = LocalOutlierFactor(n_neighbors=int(n_samples * expected_anomaly_fraction))
fig, ax = plt.subplots()
for model_idx, (linestyle, preprocessor) in enumerate(
zip(linestyles, preprocessor_list)
):
model = make_pipeline(preprocessor, lof)
model.fit(X)
y_pred = model[-1].negative_outlier_factor_
display = RocCurveDisplay.from_predictions(
y,
y_pred,
pos_label=pos_label,
name=str(preprocessor).split("(")[0],
ax=ax,
plot_chance_level=(model_idx == len(preprocessor_list) - 1),
chance_level_kw={"linestyle": (0, (1, 10))},
linestyle=linestyle,
linewidth=2,
)
ax.set_title(
"Fixed n_neighbors with varying preprocessing\non cardiotocography dataset"
)
plt.show()
Total running time of the script: (0 minutes 19.682 seconds)
Related examples